16.5 Régulation post-transcriptionnelle des gènes eucaryotes
Objectifs d’apprentissage
À la fin de cette section, vous serez en mesure de faire ce qui suit :
- Comprendre l’épissage de l’ARN et expliquer son rôle dans la régulation de l’expression génique
- Décrire l’importance de la stabilité de l’ARN dans la régulation des gènes
L’ARN est transcrit, mais doit être transformé en une forme mature avant que la traduction ne puisse commencer. Ce traitement qui a lieu après la transcription d’une molécule d’ARN, mais avant qu’elle ne soit traduite en protéine, est appelé modification post-transcriptionnelle. Comme pour les étapes épigénétiques et transcriptionnelles du traitement, cette étape post-transcriptionnelle peut également être régulée pour contrôler l’expression génique dans la cellule. Si l’ARN n’est pas modifié, transporté ou traduit, aucune protéine ne sera synthétisée.
L’épissage de l’ARN, première étape du contrôle post-transcriptionnel
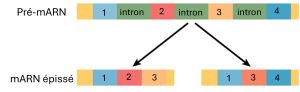
Dans les cellules eucaryotes, la transcription de l’ARN contient souvent des régions, appelées introns, qui sont supprimées avant la traduction. Les régions de l’ARN qui codent pour les protéines sont appelées exons. (figure 16.11). Après la transcription d’une molécule d’ARN, mais avant qu’elle ne quitte le noyau pour être traduite, l’ARN est modifié et les introns sont éliminés par épissage. L’épissage est effectué par les spliceosomes, des complexes ribonucléoprotéiques capables de reconnaître les deux extrémités de l’intron, de couper le transcrit en ces deux points et de rassembler les exons pour la ligature.
LIEN AVEC L’ÉVOLUTION
Épissage alternatif de l’ARN
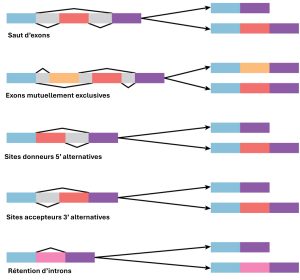
Dans les années 1970, on a observé pour la première fois des gènes présentant un épissage alternatif de l’ARN. L’épissage alternatif de l’ARN est un mécanisme qui permet de produire différents produits protéiques à partir d’un gène lorsque différentes combinaisons d’exons sont réunies pour former l’ARNm (figure 16.12). Cet épissage alternatif peut être aléatoire, mais il est plus souvent contrôlé et agit comme un mécanisme de régulation des gènes, la fréquence des différentes alternatives d’épissage étant contrôlée par la cellule comme un moyen de contrôler la production de différents produits protéiques dans différentes cellules ou à différents stades de développement. L’épissage alternatif est désormais considéré comme un mécanisme courant de régulation des gènes chez les eucaryotes ; selon une estimation, 70 % des gènes humains sont exprimés sous forme de protéines multiples par épissage alternatif. Bien qu’il existe de multiples façons d’épisser alternativement les transcriptions d’ARN, l’ordre original 5′-3′ des exons est toujours conservé. En d’autres termes, une transcription contenant des exons 1 2 3 4 5 6 7 peut être épissé 1 2 4 5 6 7 ou 1 2 3 6 7, mais jamais 1 2 5 4 3 6 7.
Comment l’épissage alternatif a-t-il pu évoluer? Les introns ont une séquence de reconnaissance de début et de fin ; il est facile d’imaginer que le mécanisme d’épissage n’identifie pas la fin d’un intron et trouve à la place la fin de l’intron suivant, supprimant ainsi deux introns et l’exon intermédiaire. En fait, il existe des mécanismes qui empêchent ce saut d’intron, mais des mutations sont susceptibles de les faire échouer. De telles « erreurs » produiraient plus que probablement une protéine non fonctionnelle. En effet, la cause de nombreuses maladies génétiques est un épissage anormal plutôt que des mutations dans une séquence codante. Toutefois, l’épissage alternatif pourrait créer une variante de la protéine sans perte de la protéine d’origine, ce qui permettrait d’adapter la nouvelle variante à de nouvelles fonctions. La duplication des gènes a joué un rôle important dans l’évolution de nouvelles fonctions d’une manière similaire en fournissant des gènes qui peuvent évoluer sans éliminer la protéine fonctionnelle d’origine.
Question : Chez le serpent des blés Pantherophis guttatus, il existe plusieurs variantes de couleurs, y compris des serpents amélaniques dont les motifs de la peau ne présentent que des pigments rouges et jaunes. La cause de l’amélanisme chez ces serpents a été récemment identifiée comme étant l’insertion d’un élément transposable dans un intron du gène OCA2 (albinisme oculocutané). Comment l’insertion de matériel génétique supplémentaire dans un intron peut-elle conduire à une protéine non fonctionnelle?
Contrôle de la stabilité de l’ARN
Avant que l’ARNm ne quitte le noyau, il reçoit deux « coiffes » protectrices qui empêchent les extrémités du brin de se dégrader au cours de son voyage. Les exonucléases 5′ et 3′ peuvent dégrader les ARN non protégés. La coiffe 5′, placée à l’extrémité 5′ de l’ARNm, est généralement composée d’une molécule de guanosine triphosphate méthylée (GTP). Le GTP est placé « en arrière » sur l’extrémité 5′ de l’ARNm, de sorte que les carbones 5′ du GTP et le nucléotide terminal sont liés par trois phosphates. La queue poly-A, qui est attachée à l’extrémité 3′, est généralement composée d’une longue chaîne d’adénine nucléotides. Ces modifications protègent les deux extrémités de l’ARN de l’attaque des exonucléases.
Une fois que l’ARN est transporté dans le cytoplasme, il est possible de contrôler la durée pendant laquelle il y réside. Chaque molécule d’ARN a une durée de vie définie et se désintègre à un rythme spécifique. Ce taux de décomposition peut influencer la quantité de protéines présentes dans la cellule. Si le taux de désintégration augmente, l’ARN n’existera pas aussi longtemps dans le cytoplasme, ce qui réduira le temps disponible pour la traduction de l’ARNm. Inversement, si le taux de désintégration est réduit, la molécule d’ARNm restera plus longtemps dans le cytoplasme et une plus grande quantité de protéines pourra être traduite. Ce taux de décroissance est appelé stabilité de l’ARN. Si l’ARN est stable, il sera détecté plus longtemps dans le cytoplasme.
La liaison des protéines à l’ARN peut également influencer sa stabilité. Des protéines appelées protéines de liaison à l’ARN, ou RBP, peuvent se lier aux régions de l’ARNm situées juste en amont ou en aval de la région codant pour la protéine. Ces régions de l’ARN qui ne sont pas traduites en protéines sont appelées régions non traduites, ou UTR. Il ne s’agit pas d’introns (ceux-ci ont été supprimés dans le noyau). Il s’agit plutôt de régions qui régulent la localisation et la stabilité de l’ARNm ainsi que la traduction des protéines. La région située juste avant la région de codage des protéines est appelée UTR 5′, tandis que la région située après la région de codage est appelée UTR 3′ (figure 16.13). La liaison des RBP à ces régions peut augmenter ou diminuer la stabilité d’une molécule d’ARN, en fonction de la RBP spécifique qui se lie.

Stabilité de l’ARN et microARN
Outre les RBP qui se lient à l’ARN et en contrôlent (augmentent ou diminuent) la stabilité, d’autres éléments appelés microARN peuvent se lier à la molécule d’ARN. Ces microARN, ou miARN, sont de courtes molécules d’ARN d’une longueur de 21 à 24 nucléotides seulement. Les miARN sont fabriqués dans le noyau sous forme de pré-miARN plus longs. Ces pré-miARN sont découpés en miARN matures par une protéine appelée Dicer. Comme les facteurs de transcription et les RBP, les miARN matures reconnaissent une séquence spécifique et se lient à l’ARN ; cependant, les miARN s’associent également à un complexe ribonucléoprotéique appelé complexe de silençage induit par l’ARN (RISC). La séquence ARN du complexe RISC s’associe à une séquence complémentaire d’un ARNm et empêche la traduction du message ou entraîne la dégradation de l’ARNm.

