15.5 Ribosomes et synthèse des protéines
Objectifs d’apprentissage
À la fin de cette section, vous serez en mesure de faire ce qui suit :
- Décrire les différentes étapes de la synthèse des protéines
- Discuter du rôle des ribosomes dans la synthèse des protéines
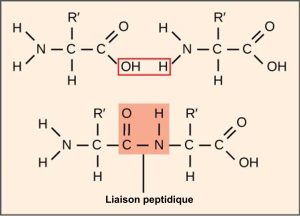
La synthèse des protéines consomme plus d’énergie que tout autre processus métabolique. À leur tour, les protéines représentent plus de masse que tout autre composant des organismes vivants (à l’exception de l’eau), et les protéines remplissent pratiquement toutes les fonctions d’une cellule. Le processus de traduction, ou synthèse des protéines, implique le décodage d’un message d’ARNm en un produit polypeptidique. Les acides aminés sont assemblés de manière covalente par des liaisons peptidiques qui s’entrecroisent sur des longueurs allant d’environ 50 à plus de 1 000 résidus d’acides aminés. Chaque acide aminé possède un groupe aminé (NH2) et un groupe carboxyle (COOH). Les polypeptides sont formés lorsque le groupe aminé d’un acide aminé forme une liaison amide (c’est-à-dire peptidique) avec le groupe carboxyle d’un autre acide aminé (figure 15.15). Cette réaction est catalysée par les ribosomes et génère une molécule d’eau.
Le mécanisme de synthèse des protéines
Outre le modèle d’ARNm, de nombreuses molécules et macromolécules contribuent au processus de traduction. La composition de chaque élément peut varier d’une espèce à l’autre ; par exemple, les ribosomes peuvent être constitués d’un nombre différent d’ARNr et de polypeptides en fonction de l’organisme. Cependant, les structures générales et les fonctions de la machinerie de synthèse des protéines sont comparables entre les bactéries et les cellules humaines. La traduction nécessite l’apport d’un modèle d’ARNm, de ribosomes, d’ARNt et de divers facteurs enzymatiques. (Remarque : Un ribosome peut être considéré comme une enzyme dont les sites de liaison des acides aminés sont spécifiés par l’ARNm).
Ribosomes
Avant même que l’ARNm ne soit traduit, la cellule doit investir de l’énergie pour construire chacun de ses ribosomes. Chez E. coli, il y a entre 10 000 et 70 000 ribosomes présents dans chaque cellule à un moment donné. Un ribosome est une macromolécule complexe composée d’ARNr structurels et catalytiques et de nombreux polypeptides distincts. Chez les eucaryotes, le nucléole est entièrement spécialisé dans la synthèse et l’assemblage des ARNr.
Les ribosomes existent dans le cytoplasme des procaryotes et dans le cytoplasme et le réticulum endoplasmique rugueux des eucaryotes. Les mitochondries et les chloroplastes possèdent également leurs propres ribosomes dans la matrice et le stroma, qui ressemblent davantage aux ribosomes procaryotes (et présentent des sensibilités médicamenteuses similaires) que les ribosomes situés juste à l’extérieur de leurs membranes externes dans le cytoplasme. Les ribosomes se dissocient en petites et grandes sous-unités lorsqu’ils ne synthétisent pas de protéines et se réassocient lors de l’initiation de la traduction. Chez E. coli, la petite sous-unité est décrite comme 30S, et la grande sous-unité comme 50S, soit un total de 70S (rappelons que les unités de Svedberg ne sont pas additives). Les ribosomes de mammifères possèdent une petite sous-unité 40S et une grande sous-unité 60S, soit un total de 80S. La petite sous-unité est responsable de la liaison de la matrice de l’ARNm, tandis que la grande sous-unité lie séquentiellement les ARNt. Chaque molécule d’ARNm est traduite simultanément par de nombreux ribosomes, qui synthétisent tous la protéine dans le même sens : lecture de l’ARNm de 5′ à 3′ et synthèse du polypeptide de la terminaison N à la terminaison C. La structure complète ARNm/polyribosome est appelée polysome.
ARNt
Les ARNt sont des molécules d’ARN structurelles qui ont été transcrites à partir des gènes par l’ARN polymérase III. Selon l’espèce, 40 à 60 types d’ARNt existent dans le cytoplasme. Les ARN de transfert servent de molécules adaptatrices. Chaque ARNt porte un acide aminé spécifique et reconnaît un ou plusieurs codons de l’ARNm qui définissent l’ordre des acides aminés dans une protéine. Les ARNt aminoacylés se lient au ribosome et ajoutent l’acide aminé correspondant à la chaîne polypeptidique. Les ARNt sont donc les molécules qui « traduisent » le langage de l’ARN en langage des protéines.
Sur les 64 codons possibles de l’ARNm – ou combinaisons de triplets A, U, G et C – trois spécifient l’arrêt de la synthèse des protéines et 61 spécifient l’ajout d’acides aminés à la chaîne polypeptidique. Parmi ces 61 codons, un codon (AUG) code également pour l’initiation de la traduction. Chaque anticodon de l’ARNt peut s’apparier avec un ou plusieurs codons de l’ARNm correspondant à son acide aminé. Par exemple, si la séquence CUA se trouve sur une matrice d’ARNm dans le cadre de lecture appropriée, elle se lie à un ARNt de leucine exprimant la séquence complémentaire, GAU. La capacité de certains ARNt à correspondre à plus d’un codon est à l’origine de la structure en blocs du code génétique.
En tant que molécules adaptatrices de la traduction, il est surprenant que les ARNt puissent intégrer autant de spécificité dans un si petit paquet. Considérons que les ARNt doivent interagir avec trois facteurs : 1) ils doivent être reconnus par la bonne aminoacyl-synthétase (voir ci-dessous) ; 2) ils doivent être reconnus par les ribosomes ; et 3) ils doivent se lier à la bonne séquence de l’ARNm.
Aminoacyl-ARNt synthétase
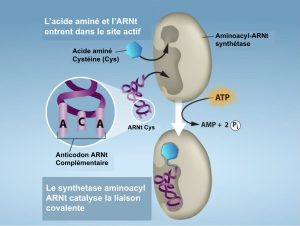
Le processus de synthèse du pré-ARNt par l’ARN polymérase III ne crée que la partie ARN de la molécule adaptatrice. L’acide aminé correspondant doit être ajouté plus tard, une fois que l’ARNt est traité et exporté vers le cytoplasme. Grâce au processus de « chargement » de l’ARNt, chaque molécule d’ARNt est reliée à l’acide aminé correct par l’une des enzymes appelées aminoacyl-ARNt synthétases. Il existe au moins un type d’aminoacyl ARNt synthétase pour chacun des 20 acides aminés ; le nombre exact d’aminoacyl ARNt synthétases varie selon les espèces. Ces enzymes lient et hydrolysent d’abord l’ATP pour catalyser une liaison à haute énergie entre un acide aminé et l’adénosine monophosphate (AMP) ; une molécule de pyrophosphate est expulsée lors de cette réaction. L’acide aminé activé est ensuite transféré à l’ARNt et l’AMP est libérée. Le terme « charge » est approprié, puisque la liaison à haute énergie qui attache un acide aminé à son ARNt est ensuite utilisée pour conduire la formation de la liaison peptidique. Chaque ARNt porte le nom de son acide aminé.
Le mécanisme de la synthèse des protéines
Comme pour la synthèse de l’ARNm, la synthèse des protéines peut être divisée en trois phases : l’initiation, l’élongation et la terminaison. Le processus de traduction est similaire chez les procaryotes et les eucaryotes. Nous examinerons ici comment la traduction se produit chez E. coli, un procaryote représentatif, et nous préciserons les différences entre la traduction procaryote et la traduction eucaryote.
Début de la traduction
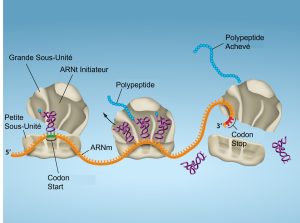
La synthèse des protéines commence par la formation d’un complexe d’initiation. Chez E. coli, ce complexe comprend le petit ribosome 30S, la matrice d’ARNm, trois facteurs d’initiation (IFs ; IF-1, IF-2, et IF-3), et un ARNt initiateur spécial, appelé ARNtfMet.
Dans l’ARNm d’E. coli, une séquence en amont du premier codon AUG, appelée séquence Shine-Dalgarno (AGGAGG), interagit avec les molécules d’ARNr qui composent le ribosome. Cette interaction ancre la sous-unité ribosomale 30S au bon endroit sur la matrice de l’ARNm. Le guanosine triphosphate (GTP), qui est un nucléotide purique triphosphate, sert de source d’énergie pendant la traduction, à la fois au début de l’élongation et pendant la translocation du ribosome. La liaison de l’ARNm au ribosome 30S nécessite également l’IF-3.
L’ARNt initiateur interagit alors avec le codon initiateur AUG. Cet ARNt transporte l’acide aminé méthionine, qui est formylé après son attachement à l’ARNt. La formylation crée une « fausse » liaison peptidique entre le groupe carboxyle du formyle et le groupe aminé de la méthionine. La liaison du fMet-ARNtfMet est médiée par le facteur d’initiation IF-2. La fMet commence chaque chaîne polypeptidique synthétisée par E. coli, mais elle est généralement supprimée une fois la traduction terminée. Lorsqu’un AUG est rencontré pendant l’élongation de la traduction, une méthionine non formylée est insérée par un Met-ARNtMet régulier. Après la formation du complexe d’initiation, la petite sous-unité ribosomique 30S est rejointe par la grosse sous-unité 50S pour former le complexe de traduction. Chez les eucaryotes, un complexe d’initiation similaire se forme, comprenant l’ARNm, la petite sous-unité ribosomique 40S, les IF eucaryotes et les nucléosides triphosphates (GTP et ATP). La méthionine sur l’ARNt initiateur chargé, appelée Met-ARNti, n’est pas formylée. Cependant, le Met-ARNti se distingue des autres Met-ARNt par le fait qu’il peut se lier aux IF.
Au lieu de se déposer sur la séquence Shine-Dalgarno, le complexe d’initiation eucaryote reconnaît la coiffe de 7-méthylguanosine à l’extrémité 5′ de l’ARNm. Une protéine de liaison à la coiffe (CBP) et plusieurs autres IF assistent le mouvement du ribosome vers la coiffe 5′. Une fois arrivé à la coiffe, le complexe d’initiation suit l’ARNm dans le sens 5′ à 3′, à la recherche du codon de départ AUG. De nombreux ARNm eucaryotes sont traduits à partir du premier AUG, mais ce n’est pas toujours le cas. Selon les règles de Kozak, les nucléotides entourant l’AUG indiquent s’il s’agit du bon codon de départ. Les règles de Kozak stipulent que la séquence consensus suivante doit apparaître autour de l’AUG des gènes de vertébrés : 5′-gccRccAUGG-3′. Le R (pour purine) indique un site qui peut être A ou G, mais qui ne peut pas être C ou U. En fait, plus la séquence est proche de ce consensus, plus l’efficacité de la traduction est élevée.
Une fois l’AUG approprié identifié, les autres protéines et la CBP se dissocient, et la sous-unité 60S se lie au complexe composé du Met-ARNti, de l’ARNm et de la sous-unité 40S. Cette étape complète l’initiation de la traduction chez les eucaryotes.
Traduction, élongation et terminaison
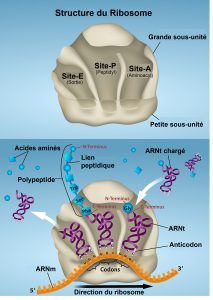
Chez les procaryotes et les eucaryotes, les bases de l’élongation sont les mêmes, c’est pourquoi nous examinerons l’élongation du point de vue d’E. coli. Lorsque le complexe de traduction est formé, la région de liaison de l’ARNt du ribosome se compose de trois compartiments. Le site A (aminoacyl) lie les ARNt aminoacyl chargés entrants. Le site P (peptidyl) lie les ARNt chargés portant des acides aminés qui ont formé des liaisons peptidiques avec la chaîne polypeptidique en croissance mais qui ne se sont pas encore dissociés de leur ARNt correspondant. Le site E (sortie ou exit) libère les ARNt déchargés (ou vide) afin qu’ils puissent être rechargés en acides aminés libres. Le méthionyl-ARNt initiateur occupe cependant le site P au début de la phase d’élongation de la traduction, tant chez les procaryotes que chez les eucaryotes.
Au cours de l’élongation de la traduction, le modèle d’ARNm fournit une spécificité de liaison à l’ARNt. Au fur et à mesure que le ribosome se déplace le long de l’ARNm, chaque codon de l’ARNm est enregistré et la liaison spécifique avec l’anticodon de l’ARNt chargé correspondant est assurée. Si l’ARNm n’était pas présent dans le complexe d’élongation, le ribosome lierait les ARNt de manière non spécifique et aléatoire.
L’élongation se déroule avec des ARNt chargés qui entrent et sortent séquentiellement du ribosome à mesure que chaque nouvel acide aminé est ajouté à la chaîne polypeptidique. Le déplacement d’un ARNt du site A à P à E est induit par des changements de conformation qui font avancer le ribosome de trois bases dans la direction 3′. L’énergie nécessaire à chaque étape du ribosome est fournie par des facteurs d’élongation qui hydrolysent le GTP. L’énergie du GTP est nécessaire à la fois pour la liaison d’un nouvel aminoacyl-ARNt au site A et pour sa translocation vers le site P après la formation de la liaison peptidique. Des liaisons peptidiques se forment entre le groupement aminé de l’acide aminé attaché à l’ARNt du site A et le groupe carboxyle de l’acide aminé attaché à l’ARNt du site P. La formation de chaque liaison peptidique est catalysée par la peptidyltransférase, une enzyme à base d’ARN intégrée à la sous-unité ribosomale 50S. L’énergie nécessaire à la formation de chaque liaison peptidique provient de la liaison à haute énergie reliant chaque acide aminé à son ARNt. Après la formation de la liaison peptidique, l’ARNt du site A qui contient maintenant la chaîne peptidique en croissance se déplace vers le site P, et l’ARNt du site P qui est maintenant vide se déplace vers le site E et est expulsé du ribosome (figure 15.18). Étonnamment, l’appareil de traduction d’E. coli ne prend que 0,05 seconde pour ajouter chaque acide aminé, ce qui signifie qu’une protéine de 200 acides aminés peut être traduite en seulement 10 secondes.
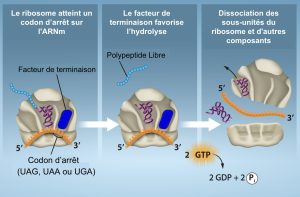
La fin de la traduction se produit lorsqu’un codon d’arrêt (ou non-sens) (UAA, UAG ou UGA) est rencontré. Lorsqu’ils s’alignent sur le site A, ces codons d’arrêt sont reconnus par des facteurs de terminaison qui ressemblent à des ARNt. Les facteurs de terminaison, tant chez les procaryotes que chez les eucaryotes, ordonnent à la peptidyltransférase d’ajouter une molécule d’eau à l’extrémité carboxyle du site P de l’acide aminé. Cette réaction force l’acide aminé du site P à se détacher de son ARNt, et la protéine nouvellement fabriquée est libérée. Les petites et grandes sous-unités ribosomiques se dissocient de l’ARNm et les unes des autres ; elles sont recrutées presque immédiatement dans un autre complexe d’initiation de la traduction. Après que de nombreux ribosomes aient terminé la traduction, l’ARNm est dégradé afin que les nucléotides puissent être réutilisés dans une autre réaction de transcription.
Modifications post-traductionnelles et ciblage des protéines
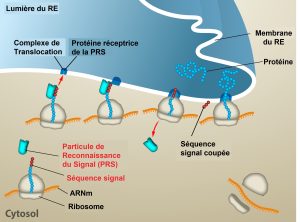
Pendant et après la traduction, les acides aminés individuels peuvent être modifiés chimiquement, des séquences de signaux peuvent être ajoutées et la nouvelle protéine peut être « repliée » dans une structure tridimensionnelle distincte à la suite d’interactions intramoléculaires. Une séquence signal est une courte séquence à l’extrémité N ou C terminale d’une protéine, qui la dirige vers un compartiment cellulaire spécifique. Ces séquences peuvent être considérées comme des « codes postaux » de la protéine vers sa destination finale et sont reconnues par des protéines de reconnaissance de signaux qui agissent comme des postières. Par exemple, une séquence signal terminale spécifique dirigera une protéine vers les mitochondries ou les chloroplastes (chez les plantes). Une fois que la protéine atteint sa destination cellulaire, la séquence signal est généralement coupée.
De nombreuses protéines se replient spontanément, mais certaines d’entre elles ont besoin de molécules d’aide, appelées chaperones, pour éviter qu’elles ne s’agrègent au cours du processus compliqué de repliement. Même si une protéine est correctement spécifiée par son ARNm correspondant, elle peut prendre une conformation complètement dysfonctionnelle si des conditions anormales de température ou de pH l’empêchent de se replier correctement.