15.1 Le code génétique
Objectifs d’apprentissage
À la fin de cette section, vous serez en mesure de faire ce qui suit :
- Expliquer le « dogme central » de la synthèse ADN-protéine
- Décrire le code génétique et la manière dont la séquence de nucléotides prescrit la séquence d’acides aminés et de protéines.
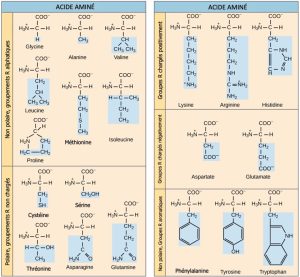
Le processus cellulaire de transcription génère l’ARN messager (ARNm), une copie moléculaire mobile d’un ou de plusieurs gènes dont l’alphabet est composé de A, C, G et d’uracile (U). Les ribosomes traduisent l’information contenu dans la séquence nucléotidiques de l’ARNm en séquences d’acides aminés, résultant en un produit protéique. C’est le dogme central de la synthèse ADN-ARN-protéine : la transcription de l’ADN en ARNm, puis la traduction de l’ARNm en protéine. Les séquences de protéines sont constituées de 20 acides aminés courants ; on peut donc dire que l’alphabet des protéines est constitué de 20 « lettres » (figure 15.2). Les différents acides aminés ont des propriétés chimiques différentes (acides ou basiques, polaires ou non polaires) et des contraintes structurelles différentes. La variation de la séquence des acides aminés est responsable de l’énorme variation de la structure et de la fonction des protéines.
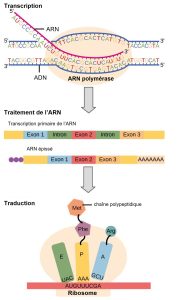
Le dogme central : L’ADN code pour l’ARN ; l’ARN code pour les protéines
Le flux d’informations génétiques dans les cellules, de l’ADN à l’ARNm puis aux protéines, est décrit par le dogme central (figure 15.3), qui stipule que les gènes spécifient la séquence des ARNm, qui à leur tour spécifient la séquence des acides aminés composant toutes les protéines. Le décodage d’une molécule en une autre est effectué par des protéines et des ARN spécifiques. L’information stockée dans l’ADN étant essentielle à la fonction cellulaire, il est intuitivement logique que la cellule fasse des copies de l’ARNm de cette information pour la synthèse des protéines, tout en gardant l’ADN lui-même intact et protégé. La copie de l’ADN en ARN est relativement simple, un nucléotide étant ajouté au brin d’ARNm pour chaque nucléotide lu dans le brin d’ADN. La traduction en protéine est un peu plus complexe, car trois nucléotides de l’ARNm correspondent à un acide aminé dans la séquence polypeptidique. Cependant, la traduction en protéine est toujours systématique et colinéaire, de sorte que les nucléotides 1 à 3 correspondent à l’acide aminé 1, les nucléotides 4 à 6 correspondent à l’acide aminé 2, et ainsi de suite.
Le code génétique est dégénéré et universel
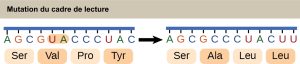
Chaque acide aminé est défini par une séquence de trois nucléotides, des triplets, appelée codon. Étant donné le nombre différent de « lettres » dans les « alphabets » de l’ARNm et des protéines, les scientifiques ont émis l’hypothèse que les acides aminés individuels devaient être représentés par des combinaisons de nucléotides. Les doublets de nucléotides ne suffiraient pas à spécifier chaque acide aminé, car il n’y a que 16 combinaisons possibles de deux nucléotides (42). En revanche, il existe 64 triplets de nucléotides possibles (43), ce qui est bien plus que le nombre d’acides aminés. Les scientifiques ont émis l’hypothèse que les acides aminés étaient codés par des triplets de nucléotides et que le code génétique était « dégénéré ». En d’autres termes, un acide aminé donné peut être codé par plus d’un triplet de nucléotides. Cela a été confirmé expérimentalement par la suite : Francis Crick et Sydney Brenner ont utilisé le mutagène chimique proflavine pour insérer un, deux ou trois nucléotides dans le gène d’un virus. Lorsqu’un ou deux nucléotides sont insérés, les protéines normales ne sont pas produites. Lorsque trois nucléotides ont été insérés, la protéine a été synthétisée et a fonctionné. Cela démontre que les acides aminés doivent être spécifiés par des groupes de trois nucléotides. Ces triplets de nucléotides sont appelés codons. L’insertion d’un ou deux nucléotides modifie complètement le cadre de lecture du triplet, altérant ainsi le message pour chaque acide aminé suivant (figure 15.5). Bien que l’insertion de trois nucléotides ait entraîné l’insertion d’un acide aminé supplémentaire pendant la traduction, l’intégrité du reste de la protéine a été maintenue.
Les scientifiques ont minutieusement résolu le code génétique en traduisant des ARNm synthétiques in vitro et en séquençant les protéines qu’ils spécifiaient (figure 15.4).
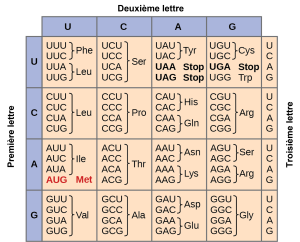
Outre les codons qui commandent l’ajout d’un acide aminé spécifique à une chaîne polypeptidique, trois des 64 codons mettent fin à la synthèse des protéines et libèrent le polypeptide de la machinerie de traduction. Ces triplets sont appelés codons d’arrêt ou codons de terminaison or non-sens. Un autre codon, AUG, a également une fonction particulière. En plus de spécifier l’acide aminé méthionine, il sert également de codon initiateur pour initier la traduction. Le cadre de lecture pour la traduction est défini par le codon initiateur AUG, situé à l’extrémité 5′ de l’ARNm. Après le codon initiateur, l’ARNm est lu par groupes de trois jusqu’à ce qu’un codon de terminaison soit rencontré.
La disposition du tableau des codons révèle la structure du code. Il existe seize « blocs » de codons, chacun étant spécifié par le premier et le deuxième nucléotides des codons à l’intérieur du bloc, par exemple le bloc « AC* » qui correspond à l’acide aminé thréonine (Thr). Certains blocs sont divisés en une moitié pyrimidine, dans laquelle le codon se termine par U ou C, et une moitié purine, dans laquelle le codon se termine par A ou G. Certains acides aminés reçoivent un bloc entier de quatre codons, comme l’alanine (Ala), la thréonine (Thr) et la proline (Pro). Certains obtiennent la moitié pyrimidine de leur bloc, comme l’histidine (His) et l’asparagine (Asn). D’autres obtiennent la moitié purine de leur bloc, comme le glutamate (Glu) et la lysine (Lys). Notez que certains acides aminés reçoivent un bloc et un demi-bloc pour un total de six codons.
La spécification d’un seul acide aminé par plusieurs codons similaires est appelée « dégénérescence ». La dégénérescence serait un mécanisme cellulaire visant à réduire l’impact négatif des mutations aléatoires. Les codons qui spécifient le même acide aminé ne diffèrent généralement que par un nucléotide. En outre, les acides aminés dont les chaînes latérales sont chimiquement similaires sont codés par des codons similaires. Par exemple, l’aspartate (Asp) et le glutamate (Glu), qui occupent le bloc GA*, sont tous deux chargés négativement. Cette nuance du code génétique garantit qu’une mutation de substitution d’un seul nucléotide peut spécifier le même acide aminé mais n’avoir aucun effet ou spécifier un acide aminé similaire, empêchant la protéine d’être rendue complètement non fonctionnelle.
Le code génétique est presque universel. À quelques exceptions près, pratiquement toutes les espèces utilisent le même code génétique pour la synthèse des protéines. La conservation des codons signifie qu’un ARNm purifié codant pour la protéine de la globine du cheval pourrait être transféré dans une cellule de tulipe, et la tulipe synthétiserait la globine du cheval. Le fait qu’il n’y ait qu’un seul code génétique est une preuve puissante que toutes les formes de vie sur Terre ont une origine commune, surtout si l’on considère qu’il existe environ 1 084 combinaisons possibles de 20 acides aminés et de 64 codons triplets.
LIEN AVEC LA MÉTHODE SCIENTIFIQUE
Lien avec la méthode scientifique
Qui a le plus d’ADN : Un kiwi ou une fraise?

Question: Est-ce qu’un kiwi et une fraise d’environ même taille (figure 15.6) auraient également à peu près la même quantité d’ADN?
Contexte: Les gènes sont portés par les chromosomes et sont constitués d’ADN. Tous les mammifères sont diploïdes, c’est-à-dire qu’ils possèdent deux copies de chaque chromosome. Cependant, toutes les plantes ne sont pas diploïdes. La fraise commune est octoploïde (8n) et le kiwi cultivé est hexaploïde (6n). Recherchez le nombre total de chromosomes dans les cellules de chacun de ces fruits et réfléchissez à la manière dont cela pourrait correspondre à la quantité d’ADN dans les noyaux cellulaires de ces fruits. Quels autres facteurs peuvent contribuer à la quantité totale d’ADN dans un seul fruit? Lisez la technique d’isolement de l’ADN pour comprendre comment chaque étape du protocole d’isolement permet de libérer et de précipiter l’ADN.
Hypothèse : Les fraises possèdent plus d’ADN que le kiwis. (Formuler l’hypothèse inverse, ie les kiwis ont plus d’ADN que le fraises serait aussi correct. Une hypothèse est formulée de façon à pouvoir y répondre oui ou non/vrai ou faux).
Testez votre hypothèse: Isolez l’ADN d’une fraise et d’un kiwi de taille similaire pour comparer leur quantité d’ADN. Réalisez l’expérience au moins en trois exemplaires pour chaque fruit.
- Préparez une bouteille de tampon d’extraction d’ADN à partir de 900 ml d’eau, 50 ml de détergent pour vaisselle et deux cuillères à café de sel de table. Mélangez par inversion (boucher le couvercle et le retourner plusieurs fois).
- Broyez une fraise et un kiwi à la main dans un sac en plastique, ou à l’aide d’un mortier et d’un pilon, ou encore avec un bol en métal et l’extrémité d’un instrument émoussé. Broyez pendant au moins deux minutes par fruit.
- Ajoutez 10 ml de tampon d’extraction d’ADN à chaque fruit et mélangez bien pendant au moins une minute.
- Éliminez les débris cellulaires en filtrant chaque mélange de fruits à travers une étamine ou un tissu poreux et dans un entonnoir placé dans une éprouvette ou un récipient approprié.
- Versez de l’éthanol ou de l’isopropanol (alcool à friction) glacé dans le tube à essai. Vous devriez observer de l’ADN blanc précipité.
- Rassemblez l’ADN de chaque fruit en l’enroulant autour de tiges de verre séparées.
Notez vos observations : Comme vous ne mesurez pas quantitativement le volume d’ADN, vous pouvez noter pour chaque essai si les deux fruits ont produit les mêmes quantités d’ADN ou des quantités différentes observées à l’œil nu. Si l’un ou l’autre fruit a produit sensiblement plus d’ADN, notez-le également. Déterminez si vos observations sont cohérentes avec plusieurs morceaux de chaque fruit.
Analysez vos données : Avez-vous remarqué une différence évidente dans la quantité d’ADN produite par chaque fruit? Vos résultats sont-ils reproductibles?
Tirez une conclusion: Compte tenu de ce que vous savez du nombre de chromosomes dans chaque fruit, pouvez-vous conclure que le nombre de chromosomes est nécessairement lié à la quantité d’ADN? Pouvez-vous identifier les inconvénients de cette procédure? Si vous aviez accès à un laboratoire, comment pourriez-vous normaliser votre comparaison et la rendre plus quantitative?

