14.4 Réplication de l’ADN chez les procaryotes
Objectifs d’apprentissage
À la fin de cette section, vous serez en mesure de faire ce qui suit :
- Expliquer le processus de réplication de l’ADN chez les procaryotes
- Discuter du rôle de différentes enzymes et protéines dans le soutien de ce processus
La réplication de l’ADN a été bien étudiée chez les procaryotes, principalement en raison de la petite taille du génome et de la grande variété de mutants disponibles. E. coli a 4,6 millions de paires de bases dans un seul chromosome circulaire et tout est répliqué en environ 42 minutes, en partant d’un seul site le long du chromosome et en faisant le tour du chromosome circulaire dans les deux sens. Cela signifie qu’environ 1 000 nucléotides sont ajoutés par seconde. Ainsi, le processus est assez rapide et se déroule sans trop d’erreurs.
La réplication de l’ADN utilise un grand nombre de protéines structurelles et d’enzymes, chacune jouant un rôle essentiel au cours du processus. L’un des principaux acteurs est l’enzyme ADN polymérase, également connue sous le nom d’ADN pol, qui ajoute des nucléotides un par un à la chaîne d’ADN en croissance qui est complémentaire au brin matrice. L’ajout de nucléotides nécessite de l’énergie ; cette énergie est obtenue à partir des nucléosides triphosphates ATP, GTP, TTP et CTP. Comme l’ATP, les autres NTP (nucléosides triphosphates) sont des molécules à haute énergie qui peuvent servir à la fois de source de nucléotides d’ADN et de source d’énergie qui alimente la polymérisation. Lorsque la liaison entre les phosphates est « rompue », l’énergie libérée est utilisée pour former la liaison phosphodiester entre le nucléotide entrant et la chaîne en croissance. Chez les procaryotes, trois principaux types de polymérases sont connus : ADN pol I, ADN pol II et ADN pol III. On peut dire que l’ADN pol III est l’enzyme principale nécessaire à la synthèse de l’ADN, que l’ADN pol I est une enzyme accessoire importante, tandis que l’ADN pol II est principalement nécessaire à la réparation.
Comment la machine de réplication sait-elle par où commencer? Il s’avère qu’il existe des séquences nucléotidiques spécifiques appelées origines de réplication où la réplication commence. E. coli n’a qu’une seule origine de réplication sur son chromosome unique (comme la plupart des procaryotes). Cette origine de réplication est une séquence d’environ 245 paires de bases et est riche en séquences AT. L’origine de la réplication est reconnue par certaines protéines qui se lient à ce site. Une enzyme appelée hélicase déroule l’ADN en brisant les liaisons hydrogène entre les paires de bases azotées. L’hydrolyse de l’ATP est nécessaire pour ce processus. Au fur et à mesure que l’ADN s’ouvre, des structures en forme de Y appelées fourches de réplication se forment. Deux fourches de réplication sont formées à l’origine de la réplication et celles-ci s’étendent de manière bidirectionnelle au fur et à mesure que la réplication progresse. Les protéines de liaison simple brin enrobent les simples brins d’ADN près de la fourche de réplication pour empêcher l’ADN simple brin de se refermer en une double hélice. Les protéines de liaison simple brins protègent aussi l’ADN simple brin des nombreuses enzymes présentent dans le cytoplasme qui sont capables de digérer l’ADN simple brin.
L’ADN polymérase a deux restrictions importantes : elle n’est capable d’ajouter des nucléotides que dans la direction 5′ à 3′ (un nouveau brin d’ADN ne peut être synthétisé que dans cette direction). Elle a également besoin d’un groupe 3′-OH libre auquel elle peut ajouter des nucléotides en formant une liaison phosphodiester entre l’extrémité 3′-OH et le phosphate 5′ du nucléotide suivant. Cela signifie essentiellement qu’elle ne peut pas ajouter de nucléotides si un groupe 3′-OH libre n’est pas disponible. Alors comment ajoute-t-elle le premier nucléotide? Le problème est résolu à l’aide d’une amorce qui fournit l’extrémité libre 3′-OH. Une autre enzyme, l’ARN primase, synthétise un segment d’ARN d’environ cinq à dix nucléotides de long et complémentaire de l’ADN matrice. Parce que cette séquence amorce la synthèse de l’ADN, elle est appelée à juste titre l’amorce. L’ADN polymérase peut maintenant étendre cette amorce d’ARN, en ajoutant un par un des nucléotides complémentaires au brin matrice (Figure 14.14).
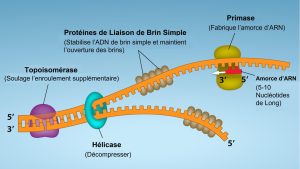
La fourche de réplication se déplace à la vitesse de 1 000 nucléotides par seconde. Une topoisomérase (ou gyrase) empêche l’enroulement excessif de la double hélice d’ADN en avant de la fourche de réplication lorsque les deux brins d’ADN sont dissocié par l’hélicase; elle le fait en provoquant des entailles temporaires dans un brin de l’ADN, puis en la refermant. Parce que l’ADN polymérase ne peut synthétiser que dans la direction 5′ à 3′, et parce que la double hélice de l’ADN est antiparallèle, il y a un léger problème à la fourche de réplication. Les deux brins d’ADN matrice ont des orientations opposées : un brin est orienté dans la direction 5′ à 3′ et l’autre est orienté dans la direction 3′ à 5′. Un seul nouveau brin d’ADN, celui qui est complémentaire au brin d’ADN parental 3′ à 5′, peut être synthétisé en continu au fur et à mesure que la fourche de réplication avance. Ce brin synthétisé en continu est connu sous le nom de brin continu. L’autre brin, complémentaire à l’ADN parental 5′ à 3′, est synthétisé loin de la fourche de réplication, en petits fragments appelés fragments d’Okazaki, chacun nécessitant une amorce pour démarrer la synthèse. De nouveaux segments d’amorce sont déposés dans la direction de la fourche de réplication, mais chacun d’entre eux est orienté dans la direction opposée à celle-ci. Les fragments d’Okazaki portent le nom du scientifique japonais qui les a découvert pour la première fois. Le brin avec les fragments d’Okazaki est connu sous le nom de brin discontinu.
Le brin continu peut être synthétisé à partir d’une seule amorce, tandis que le brin discontinu a besoin d’une nouvelle amorce pour chacun des courts fragments d’Okazaki. La direction globale du brin discontinu sera de 3′ à 5′, et celle du brin continu de 5′ à 3′. Une protéine appelée pince coulissante maintient l’ADN polymérase en place pendant qu’elle continue d’ajouter des nucléotides. La pince coulissante est une protéine en forme d’anneau qui se lie à l’ADN et maintient la polymérase en place. Au fur et à mesure de la synthèse, les amorces d’ARN sont remplacées par de l’ADN. Les amorces sont éliminées par l’activité exonucléase de l’ADN pol I, qui utilise l’ADN derrière l’ARN comme sa propre amorce et comble les lacunes laissées par l’élimination des nucléotides d’ARN et l’ajout de nucléotides d’ADN. Les brèches qui restent entre l’ADN nouvellement synthétisé (qui a remplacé l’amorce d’ARN) et l’ADN précédemment synthétisé sont scellées par l’enzyme ADN ligase, qui catalyse la formation de liaisons phosphodiester entre l’extrémité 3′-OH d’un nucléotide et l’extrémité phosphate 5′ de l’autre fragment.
Une fois le chromosome complètement répliqué, les deux copies d’ADN se déplacent dans deux cellules différentes lors de la division cellulaire.
Le processus de réplication de l’ADN peut être résumé comme suit :
- L’ADN se déroule (ou dissocie) à l’origine de la réplication.
- L’hélicase ouvre les fourches de réplication de manière bidirectionnelle.
- Les protéines de liaison simple brin enrobent l’ADN autour de la fourche de réplication pour empêcher la réassociation de l’ADN en double brin.
- La topoisomérase (ou gyrase) se lie à la région située en amont de la fourche de réplication pour empêcher le surenroulement de l’ADN.
- La primase synthétise des amorces d’ARN complémentaires au brin d’ADN.
- L’ADN polymérase III commence à ajouter des nucléotides à l’extrémité 3′-OH de l’amorce.
- L’allongement du brin continu et du brin discontinu se poursuit.
- Les amorces d’ARN sont éliminées par l’activité des exonucléases.
- L’ADN pol I ajoute les nucléotides d’ADN manquants.
- L’espace entre les deux fragments d’ADN est scellé par l’ADN ligase, qui aide à la formation de liaisons phosphodiester.
Le tableau 14.1 résume les enzymes impliquées dans la réplication de l’ADN des procaryotes et les fonctions de chacune d’entre elles.
Tableau 14.1. Réplication de l’ADN chez les procaryotes : Enzymes et leurs fonctions


