N04: Le traitement des données
Maheshwar Panday
Se familiariser avec tidyverse
Vous êtes-vous déjà demandé comment interpréter un ensemble de données ? Parfois, les ensembles de données sont disponibles mais dans des formats qui semblent déroutants ? Parfois, le format d’organisation dans lequel vous recevez un ensemble de données n’a pas beaucoup de sens et n’est pas très utile pour vous renseigner sur les données. Pour obtenir des informations sur vos données, vous devez parfois utiliser des outils d’organisation des données – en R, nous appelons cette série d’outils “data wrangling”.
En chargeant la série de paquets tidyverse – un groupe de paquets (tidyr, dplyr et ggplot2), nous pouvons facilement organiser, ordonner et visualiser nos données afin de vérifier qu’elles sont organisées dans des formats raisonnables.
Dans cet article, nous allons passer en revue quelques-unes des opérations de base de tidyverse qui sont parmi les plus utilisées, et les utiliser pour manipuler un ensemble de données catégoriques afin de présenter l’information de manière succincte.
Quelques ressources pratiques : voici quelques feuilles de contrôle pour les notions de base pour le nettoyage et le traitement des données dans RStudio en utilisant la série de paquets tidyverse (N.B. ces feuilles de contrôle sont développés par posit) :
Data Wrangling : https://www.rstudio.com/wp-content/uploads/2015/02/data-wrangling-cheatsheet.pdf
Tidyr : https://bioinformatics.ccr.cancer.gov/docs/rintro/resources/tidyr_cheatsheet.pdf
Dplyr : https://nyu-cdsc.github.io/learningr/assets/data-transformation.pdf
Chargement des progiciels
library(tidyverse) # core series of data wrangling packages.
library(dplyr) # core data wrangling grammar
library(ggplot2) # data visualisation tools
library(RColorBrewer) # colour palettes
library(here) # file directories
library(gridExtra) # arranging plots Les données utilisées pour cette série d’exercices tidyverse proviennent du dépôt de données d’apprentissage automatique de l’Université de Californie à Irvine (UC Irvine Machine learning). Des informations sommaires sur l’ensemble de données et les variables qu’il contient sont disponibles sur le lien suivant : https://archive.ics.uci.edu/dataset/915/differentiated+thyroid+cancer+recurrence
D’autres informations sur l’ensemble de données et son utilisation sont disponibles dans le document source
Borzooei, S., Briganti, G., Golparian, M. et al. Machine learning for risk stratification of thyroid cancer patients: a 15-year cohort study. Eur Arch Otorhinolaryngol (2023). https://doi.org/10.1007/s00405-023-08299-w
Chargement d’un ensemble de données et comprendre son format d’organisation
Chargez ci-dessous une copie de l’ensemble de données sur le cancer de la thyroïde et imprimez l’en-tête (les 6 premières rangées).
thyroid.data <- read.csv("Thyroid_Diff.csv")
print (head(thyroid.data))
export.path <- here::here("/Tidyverse_DataWrangling")Que signifie un cadre de données large, long et ordonné ? Pourquoi les formats d’ensemble de données ont-ils de l’importance ?
Les cadres de données ont une structure définie et une certaine terminologie est utilisée pour décrire les différentes structures que peuvent prendre les cadres de données.
- Les cadres de données sont considérés comme BIEN RANGÉS lorsque chaque ligne est un cas pour lequel des observations sont faites dans les colonnes.
- Les cadres de données sont considérés comme LARGES lorsqu’il y a plus de colonnes que de lignes.
- Les cadres de données sont considérés comme LONGS lorsqu’il y a plus de lignes que de colonnes.
Si vous regardez le cadre de données sur le cancer de la thyroïde, le cadre de données est-il ordonné ? Le cadre de données est-il long ou large ?
Pour plus d’informations sur la manipulation des données et les opérations à travers le tidyverse, vous pouvez consulter ce chapitre du livre en ligne R for Data Science : 2e (dont certaines informations ont été utilisées pour construire ces activités) : https://r4ds.hadley.nz/data-transform
view(thyroid.data)
# the dataframe is tidy and in a wide format - wrangling will be needed to present informative counts from within the dataframe Comment puis-je obtenir des informations sur mes données à partir des données dont je dispose ?
Lorsque vous regardez le cadre de données thyroid.data, chaque colonne décrit quelque chose sur chaque patient et sur le cancer de la thyroïde qui lui est associé. Mais comment interpréter les tendances ou visualiser facilement les informations contenues dans le cadre de données ? Pour ce faire, vous devrez faire un peu de ce que nous appelons le traitement des données. Il s’agit d’organiser et de modifier la structure de votre cadre de données afin de présenter des informations pertinentes par le biais de statistiques ou de visualisations succinctes et ciblées.
Si vous imprimez les noms des colonnes, vous verrez qu’il y a beaucoup de caractéristiques catégorielles différentes qui décrivent le patient et son cancer. Comment visualiser des données catégorielles de manière à présenter des nombres ou des proportions basés sur ces variables catégorielles ? Cette activité de manipulation de données vous aidera à y parvenir. En route pour le tidyverse ! 🙂 .
colnames ( thyroid.data)Comprendre l’opérateur pipe %>% et comment l’utiliser pour pouvoir manier vos données
La première chose à comprendre est l’opérateur pipe %>%. Ce petit champion de la manipulation de données vous permet d’écrire proprement et d’enchaîner une série d’opérations de manipulation de données afin d’effectuer de manière transparente une série de manipulations de données pour produire la structure souhaitée de l’image de données avec les colonnes et les lignes nécessaires pour produire les visualisations qui présentent le mieux les informations contenues dans l’image de données.
Tout d’abord, l’opérateur pipe peut être appliqué aux vecteurs ainsi qu’aux cadres de données. De la même manière que nous pouvons “canaliser” les sorties de vecteurs dans des opérations, nous pouvons “canaliser” des colonnes ou des cadres de données entiers dans des fonctions de manipulation de données afin de produire la structure de données requise.
Pour commencer la prochaine série de questions, vous devrez être à l’aise avec l’opérateur pipe pour manipuler vos données à partir du cadre de données sur le cancer de la thyroïde que vous avez chargé dans RStudio. La première activité consiste à manipuler le cadre de données pour compter le nombre de cas de cancer de la thyroïde pour chacune des quatre pathologies différentes de l’ensemble de données.
## ------------------------------------------------------- ##
#### Using the pipe operator to pass inputs to functions ####
## ------------------------------------------------------- ##
# give this code and make it visible to the students
# try computing the mean of a vector of numbers :
no.piped.mean <- mean(c(1,2,3,4,5,6,7,8,9, 10, 11, 12))
piped.mean <- c(1,2,3,4,5,6,7,8,9,10,11, 12) %>% mean()
print (paste("mean without pipe operator: ", no.piped.mean,
"mean with pipe operator: ", piped.mean))
## --------- wrangling the thyroid cancer dataframe ---------- ##
## --------------------------------------------- ##
##### 1. grouping thyroid cancer by pathology #####
## --------------------------------------------- ##
# this is the first step to organizing the dataframe for downstream analysis
# the pipe operator is taking the thyroid.data dataframe and applying the group_by function to it grouping rows together based on the values in the pathology column of the dataframe.
thyroid.by.pathology <- thyroid.data %>% group_by(Pathology)
print (head(thyroid.by.pathology))
## ----------------------------------------------- ##
##### 2. summarise the counts of each pathology #####
## ----------------------------------------------- ##
# now you can prepare a frequency table - of all the cases in this thyroid cancer dataset, how many cases are of EACH pathology?
# this step "pipes" the thyroid.by.pathology dataframe produced in the previous step, to the summarise function, counting the frequency of each pathology across the entire dataset
pathology.frequencies<- thyroid.by.pathology %>% summarise (Frequency = n())
print (head(pathology.frequencies))L’organisation visuelle des données, obtenir des renseignements sur vos données à partir de vos données
Cet ensemble de données contient 4 pathologies cancéreuses uniques : Folliculaire, Cellule de Hurthel, Micropapillaire et Papillaire. Combien de cas de chaque type se trouvent dans cet ensemble de données ? Présentez vos résultats sous la forme d’un histogramme de fréquence. Annotez les barres de l’histogramme de fréquence pour indiquer le nombre de cas dans chaque pathologie.
Conseil : utilisez la série de packages tidyverse pour manipuler vos données.
# Use dplyr to group by Pathology and count the number of occurrences
# this time all the steps outlined in the preliminary code chunk was piped together and brought together to produce a pathology frequency dataframe.
pathology_freq <- thyroid.data %>%
group_by(Pathology) %>%
summarise(Frequency = n())
# Print the frequency distribution
print(pathology_freq)
# Use ggplot2 to create a bar plot of the frequency histogram.
thyroid.cancer.freqplot <- ggplot(pathology_freq, aes(x = Pathology, y = Frequency)) +
geom_bar(stat = "identity", fill = "lightsteelblue3") +
geom_text(aes(label = Frequency), vjust = 1.5, hjust = 0.5) +
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
theme_classic()+
labs(x = "Pathology", y = "Frequency", title = "Frequency Distribution : Pathology Categories")
# plot inspection
print (thyroid.cancer.freqplot)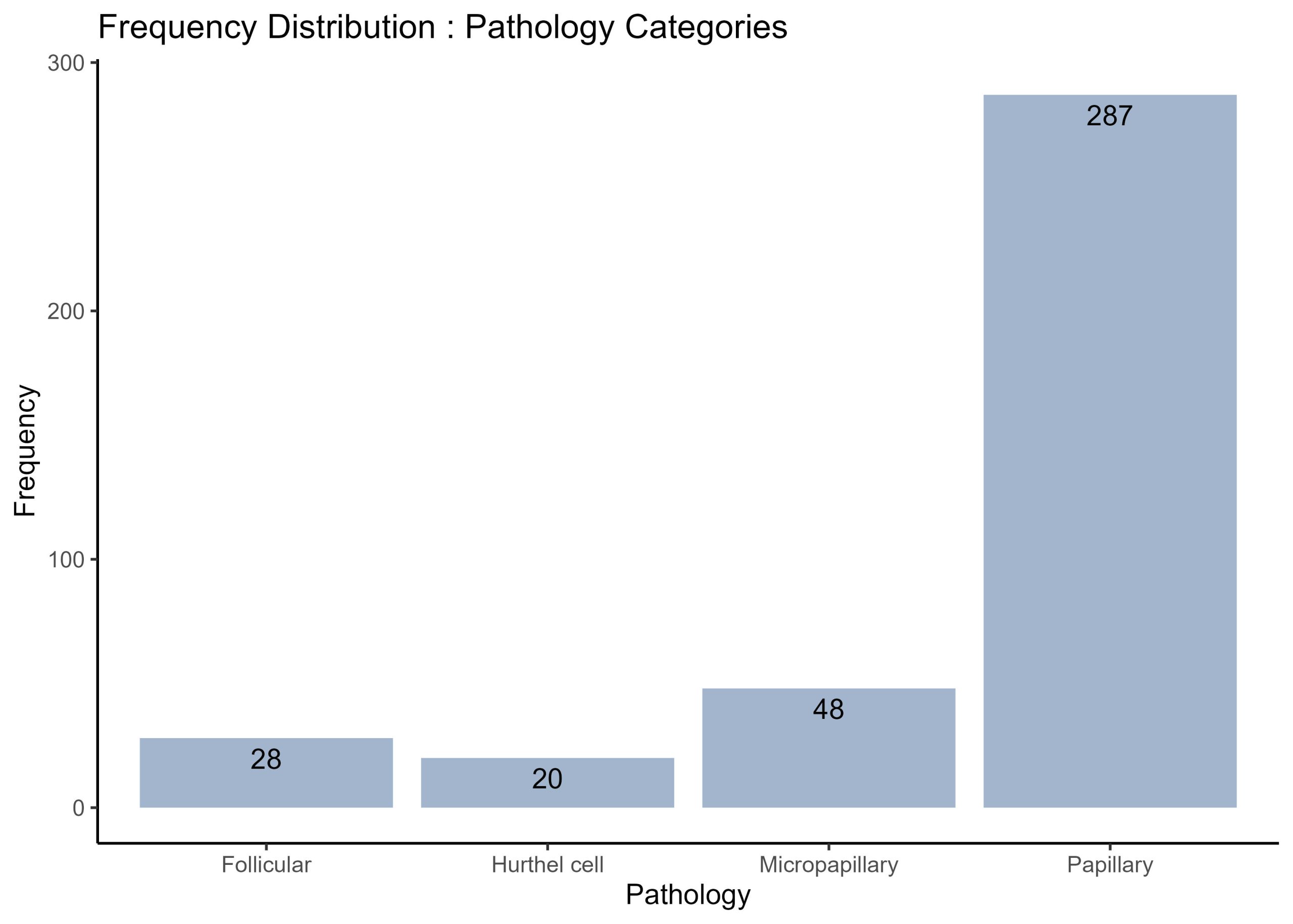
Le premier histogramme de fréquence est intéressant car il permet de visualiser facilement le nombre de cas de chaque type de pathologie cancéreuse dans l’ensemble de données, mais il est important de noter qu’il existe d’autres données sur les patients qui sont également précieuses, mais qui ne sont pas présentes dans la visualisation précédente. Supposons que vous souhaitiez également connaître le nombre d’hommes et de femmes pour chaque pathologie. Comment obtiendrez-vous les proportions d’hommes et de femmes pour chaque pathologie et comment modifieriez-vous l’histogramme de fréquence ci-dessus pour visualiser le nombre d’hommes et de femmes pour chaque groupe de pathologie ?
Modifiez l’histogramme de fréquence ci-dessus pour montrer les proportions d’hommes et de femmes dans chaque pathologie, et annotez chaque barre avec les nombres d’hommes et de femmes dans chaque groupe de pathologie.
# Group by Pathology and Gender, and count the number of occurrences
gender_pathology_freq <- thyroid.data %>%
group_by(Pathology, Gender) %>%
summarise(Frequency = n())
# Plot the stacked bar chart
thyroid.freqplot.by.Gender <- ggplot(gender_pathology_freq, aes(x = Pathology, y = Frequency, fill = Gender)) +
geom_bar(stat = "identity") +
geom_text(aes(label = Frequency), position = position_stack(vjust = 0.5)) +
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
theme_classic()+
labs(x = "Pathology", y = "Frequency", fill = "Gender", title = "Frequency Distribution of Each Pathology Category by Gender") # set the fill of each bar to show the proportions of genders
# plot inspection step
print (thyroid.freqplot.by.Gender)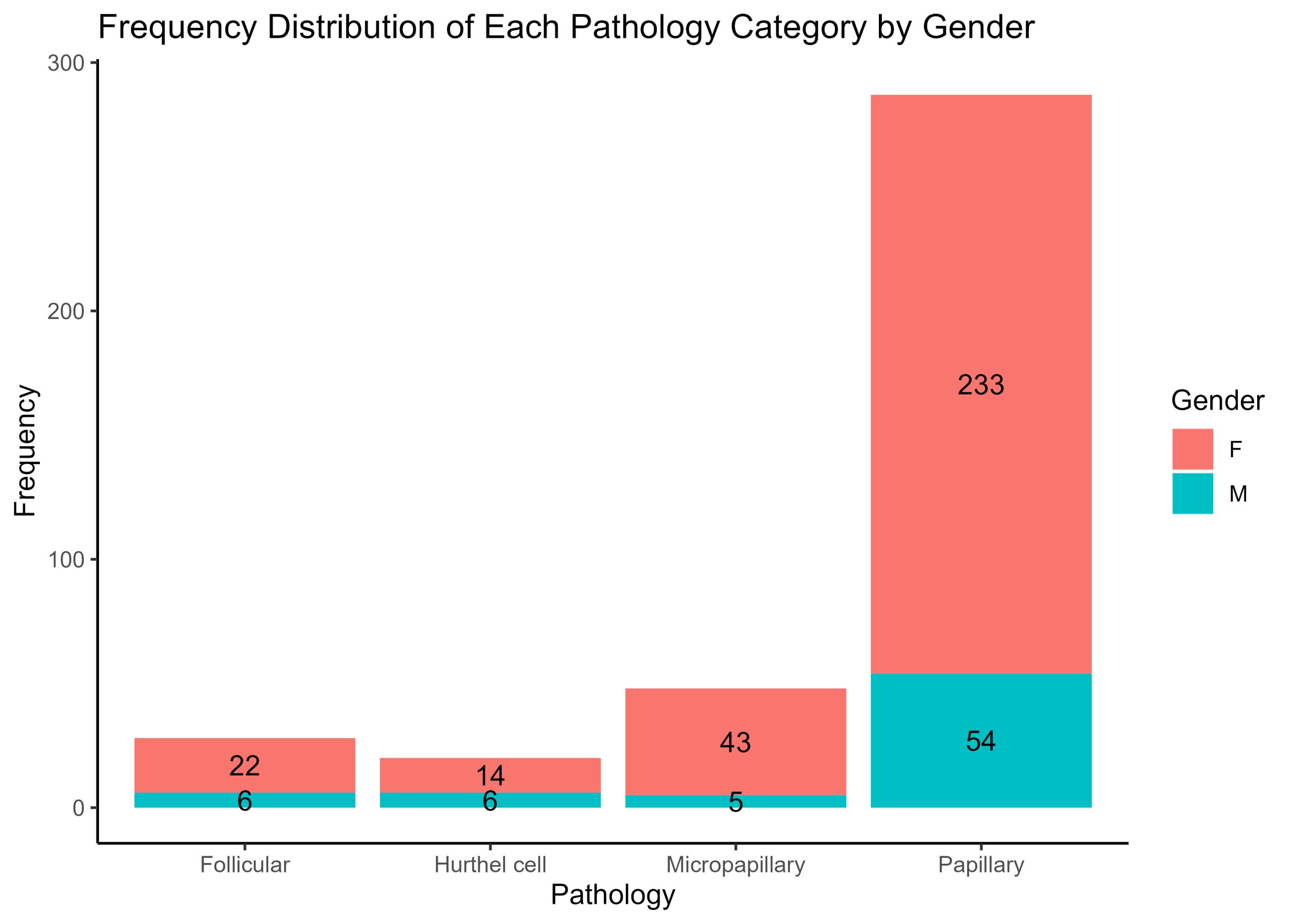
Visualiser les différentes proportions d’examens physiques pour catégorie de pathologie
Les histogrammes de fréquence sont un moyen utile de visualiser les nombres et les proportions, mais supposons que vous souhaitiez voir la proportion d’un ensemble de conditions diagnostiques dans une série de pathologies cancéreuses. Pour chaque patient, l’examen physique de la glande thyroïde a été classé dans l’une des cinq catégories suivantes : goitre diffus, goitre multinodulaire, normal, goitre nodulaire unique gauche et goitre nodulaire unique droit.
Il existe quatre pathologies distinctes (comme vous l’avez appris dans l’exercice précédent). Supposons que vous exploitiez ces données et que vous souhaitez connaître les proportions de chaque catégorie de diagnostic physique pour chacune des pathologies. Oui, vous pouvez utiliser un histogramme de fréquence comme développé précédemment, mais vous pouvez également utiliser un diagramme circulaire pour rendre les proportions des catégories de diagnostic physique facilement visibles.
Préparez une série de 4 diagrammes circulaires – un diagramme pour chaque pathologie et, dans chaque diagramme, indiquez la proportion de cas pour chaque catégorie d’examen physique.
#create a custom colour palette :
colour.palette <- c("maroon3", "mediumslateblue", "olivedrab3", "cadetblue2", "darkgoldenrod2" )
# Group by Pathology and Physical.Examination, and count the number of occurrences
pathology_exam_freq <- thyroid.data %>%
group_by(Pathology, Physical.Examination) %>%
summarise(Frequency = n())
# Calculate the total number of each Pathology
total_pathology <- pathology_exam_freq %>%
group_by(Pathology) %>%
summarise(Total = sum(Frequency))
# Join the two dataframes together
pathology_exam_freq <- left_join(pathology_exam_freq, total_pathology, by = "Pathology")
# Calculate the proportion
pathology_exam_freq <- pathology_exam_freq %>%
mutate(Proportion = Frequency / Total)
# Create a pie chart for each Pathology - to see the distribution of physical examination values
Diagnosis.by.Pathology <- pathology_exam_freq %>%
ggplot(aes(x = "", y = Proportion, fill = Physical.Examination)) +
geom_bar(width = 1, stat = "identity") + # fill each bar for a physical examination category
geom_text(aes(label = Frequency), position = position_stack(vjust = 0.5)) +
coord_polar("y", start = 0) + # create a circular histogram- pie chart
facet_wrap(~Pathology) + # this generates a series of 4 plots for each pathology
theme_void() + # aesthetics
theme(legend.position = "right") +
scale_fill_manual(values = colour.palette)+ # fill each bar with a specific colour from palette
labs(fill = "Physical Examination", title = "Proportion of Cases in Each Physical Examination Category by Pathology")
print (Diagnosis.by.Pathology)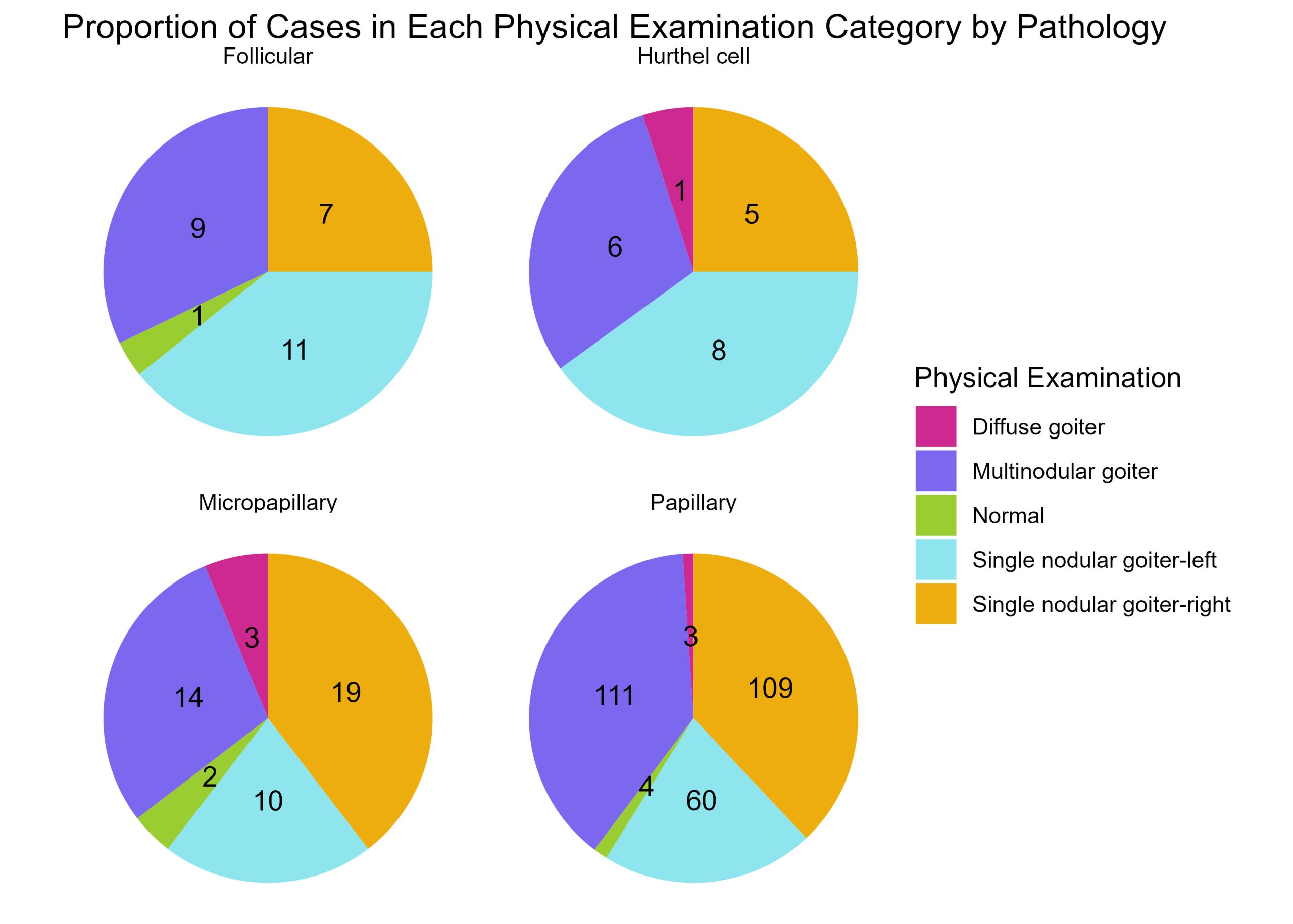
Il s’agit ci-dessus d’un bon moyen de montrer combien de caractéristiques d’examen physique sont présentes dans chaque pathologie cancéreuse. Maintenant, comme vous l’avez fait pour l’histogramme de fréquence ci-dessus, séparez ces données pour montrer les proportions des catégories d’examen physique dans chaque pathologie cancéreuse, par sexe. Cette fois, vous devez créer deux séries de 4 graphiques. Une série pour les hommes et une autre pour les femmes. Chaque graphique montre la proportion de catégories d’examens physiques pour une pathologie donnée.
#create a custom colour palette :
colour.palette <- c("maroon3", "mediumslateblue", "olivedrab3", "cadetblue2", "darkgoldenrod2" )
# Group by Gender, Pathology and Physical.Examination, and count the number of occurrences
gender_pathology_exam_freq <- thyroid.data %>%
group_by(Gender, Pathology, Physical.Examination) %>%
summarise(Frequency = n())
# Calculate the total number of each Gender and Pathology
total_gender_pathology <- gender_pathology_exam_freq %>%
group_by(Gender, Pathology) %>%
summarise(Total = sum(Frequency))
# Join the two dataframes together
gender_pathology_exam_freq <- left_join(gender_pathology_exam_freq, total_gender_pathology, by = c("Gender", "Pathology"))
# Calculate the proportion
gender_pathology_exam_freq <- gender_pathology_exam_freq %>%
mutate(Proportion = Frequency / Total)
# Create a pie chart for each Gender and Pathology
Diagnosis.by.Pathology.by.Gender<- gender_pathology_exam_freq %>%
ggplot(aes(x = "", y = Proportion, fill = Physical.Examination)) +
geom_bar(width = 1, stat = "identity") +
geom_text(aes(label = Frequency), position = position_stack(vjust = 0.5)) +
coord_polar("y", start = 0) +
facet_grid(Gender ~ Pathology) + # create a grid of plots - gender = column, pathologies = rows
theme_void() +
theme(legend.position = "top",
plot.title = element_text(hjust = 0.5)) +
scale_fill_manual(values = colour.palette)+
labs(fill = "Physical Examination", title = "Proportion of Cases for Each Physical Examination Category by Pathology and Gender")
print (Diagnosis.by.Pathology.by.Gender)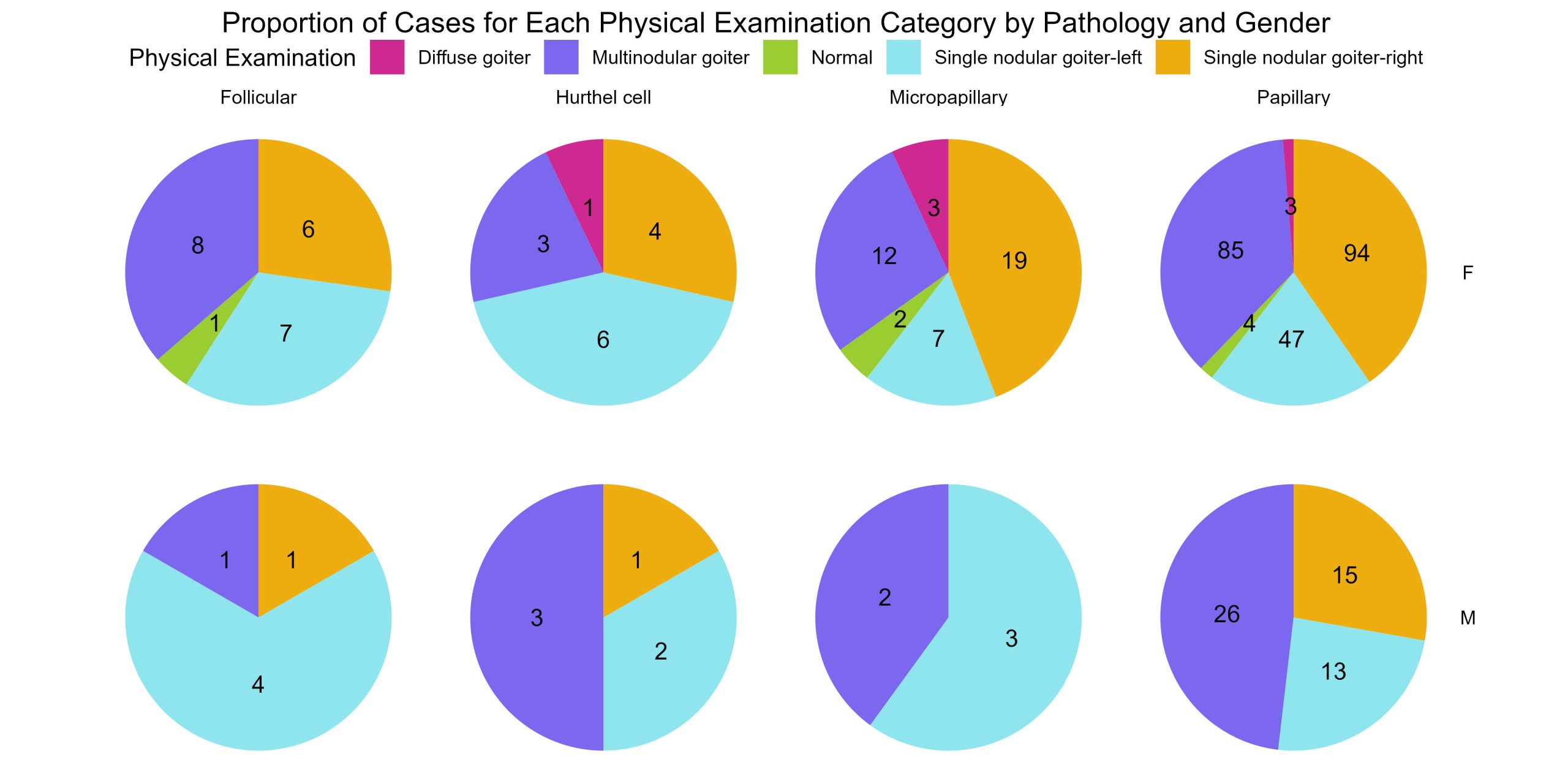
Donnez des diagrammes en boîte de l’âge par sexe pour chaque diagnostic physique par pathologie
La grammaire de manipulation des données fournie par les progiciels de base de tidyverse constitue un moyen essentiel de regrouper et d’organiser vos données afin de visualiser les tendances, les effectifs et les proportions plus simplement, au moyen de visualisations convaincantes et succinctes.
Un excellent exemple est la création d’une série de diagrammes en boîte. La série de 8 diagrammes circulaires que vous venez de générer dans le dernier exercice montre les proportions des catégories d’examen physique pour chaque pathologie et pour chaque sexe. Mais tous les patients de cet ensemble de données n’ont pas le même âge. Il serait important de connaître également les données démographiques relatives à l’âge. Créez une série de diagrammes en boîte montrant l’âge par sexe pour chaque catégorie d’examen physique pour chaque pathologie cancéreuse. Il s’agit d’une série de 20 diagrammes en boîte.
Cette fois, nous devons utiliser des diagrammes en boîte plutôt que des diagrammes circulaires ou des histogrammes de fréquence, puisque nous voulons montrer l’éventail des âges par sexe pour chaque groupe de catégories.
# Create boxplots of Age by Gender for each Physical.Examination for each Pathology
thyroid.age.boxplots <- thyroid.data %>%
ggplot(aes(x = Gender, y = Age, fill = Gender)) +
geom_boxplot() +
facet_grid(Pathology ~ Physical.Examination) + # produces a grid of plots - each row is a pathology, and each column is a physical examiantion category with boxplots of age by gender
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
labs(x = "Gender", y = "Age", fill = "Gender", title = "Boxplots of Age by Gender for Each Physical Examination Category by Pathology")
print (thyroid.age.boxplots)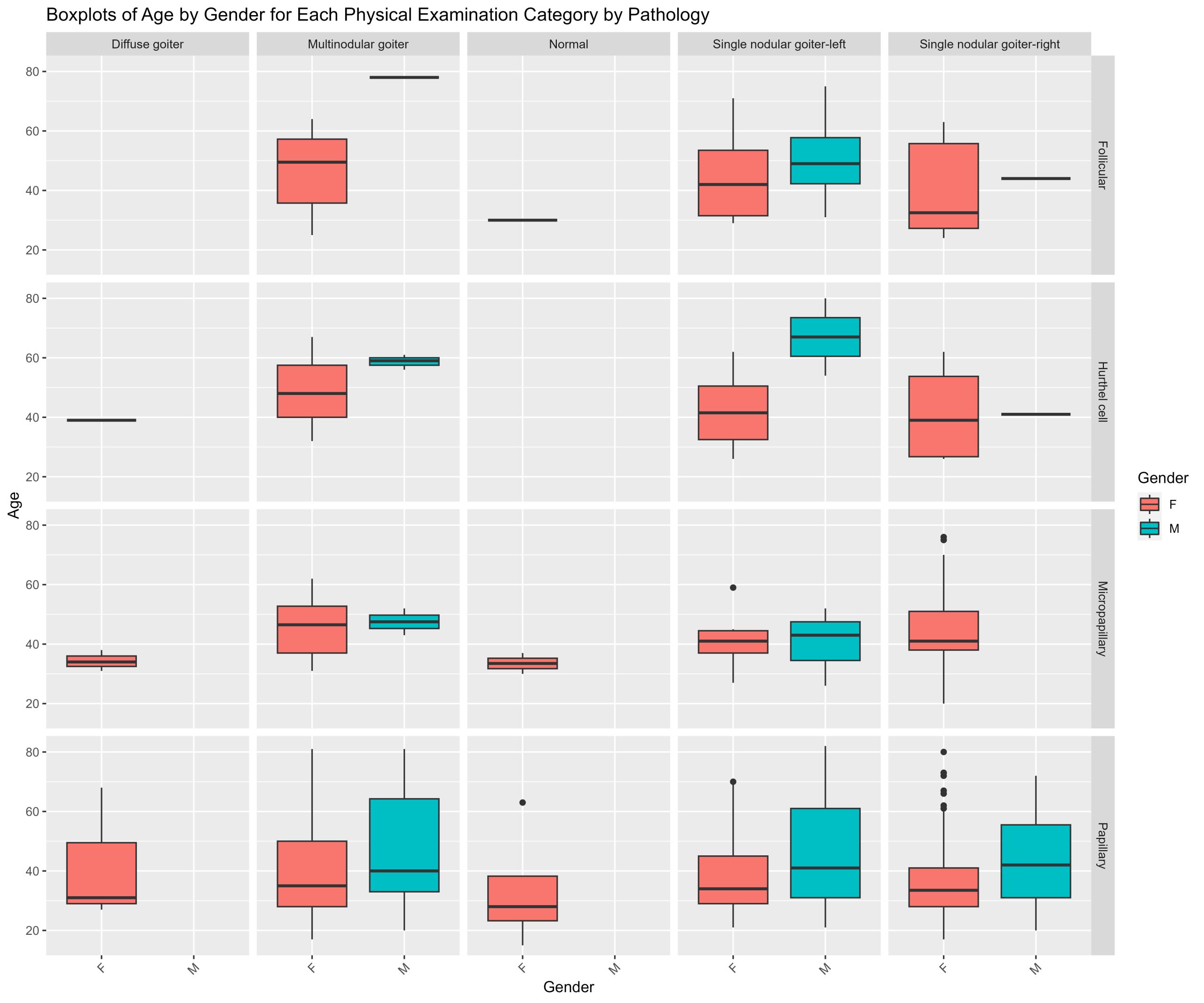
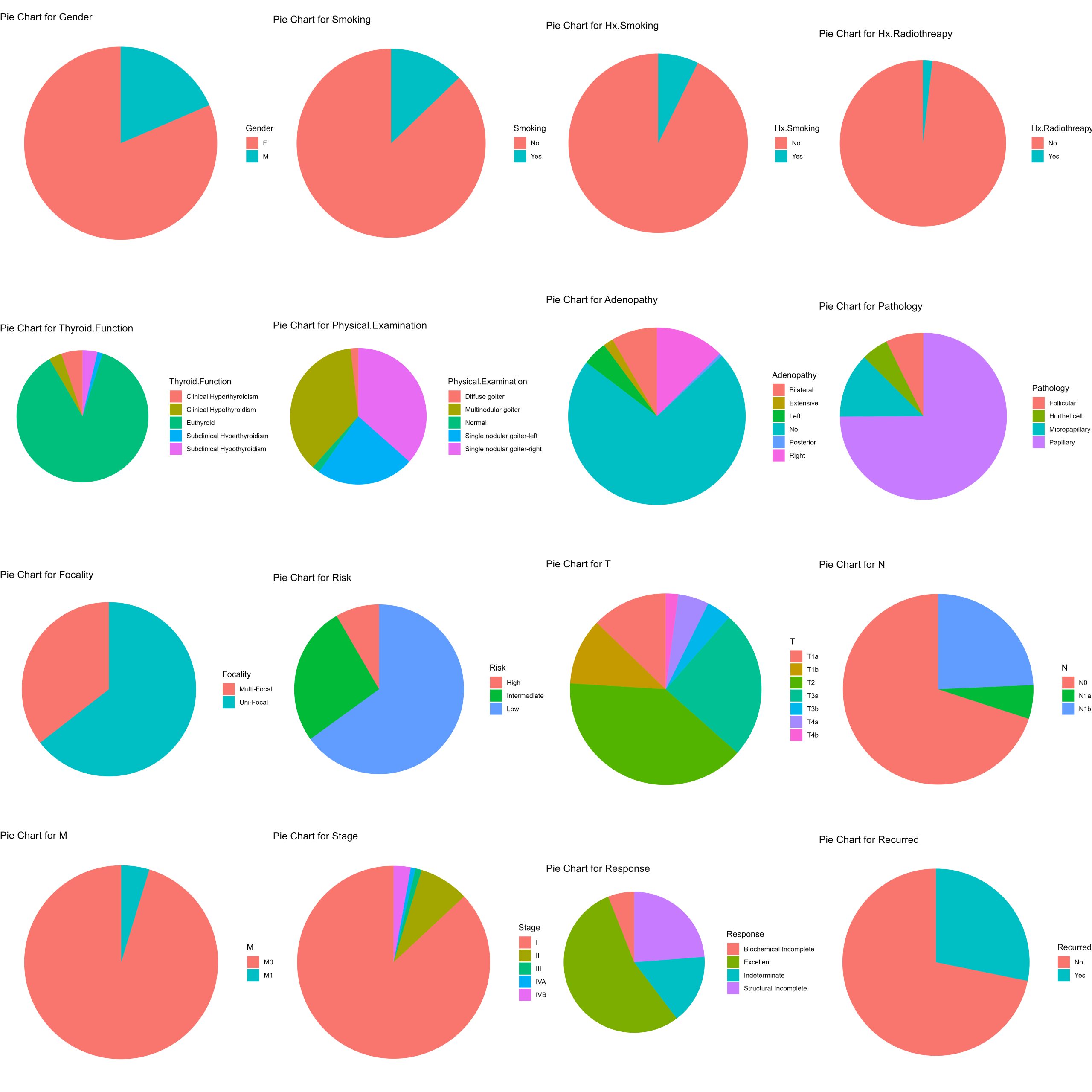
Des diagrammes circulaires pour chaque caractéristique
Bien que vous puissiez préparer une série d’effectifs, de proportions ou de diagrammes en boîte pour diverses combinaisons de caractéristiques imbriquées les unes dans les autres. Une étape essentielle consiste à comprendre quelles sont vos caractéristiques, ce que signifient les informations catégorielles contenues dans chaque caractéristique et quelles proportions de vos données se retrouvent dans chaque caractéristique. Vous pouvez y parvenir en créant des diagrammes circulaires pour chaque caractéristique, à l’exception de l’âge du patient. Vous devriez probablement effectuer cette opération au début avant d’entreprendre votre propre analyse d’un ensemble de données catégorielles, mais ce processus est un peu plus compliqué et nous le réservons donc pour la fin. Le processus implique à la fois une série d’opérations de manipulation et des outils de visualisation.
C’est à partir d’analyses exploratoires telles que les graphiques générés dans cette grille de diagrammes circulaires que le chaînage des informations sur les caractéristiques de la pathologie, du sexe, de l’examen physique et de l’âge a semblé intéressant !
# Exclude the "Age" column
data_without_age <- thyroid.data[ , !(names(thyroid.data) %in% "Age")]
# Initialize an empty list to store the plots
plot_list <- list()
## --------------------------------------------- ##
###### loop through columns - get proportions #####
###### & Generate the pie charts each column #####
## --------------------------------------------- ##
for (column_name in names(data_without_age)) {
# Calculate proportions
proportions <- data_without_age %>%
group_by(.data[[column_name]]) %>% # group by common features.
summarise(n = n()) %>% # produce summary statistics
mutate(prop = n / sum(n)) # mutate produces a column with proportions in one step
# Create pie chart
pie_chart <- ggplot(proportions, aes(x = "", y = prop, fill = .data[[column_name]])) +
geom_bar(width = 1, stat = "identity") +
coord_polar("y", start = 0) +
labs(title = paste("Pie Chart for", column_name), x = NULL, y = NULL, fill = column_name) +
theme(plot.margin = margin (1,1,1,1, "cm"))+ # common margin to each plot
theme_void() #aesthetics - blank backgrounds
# Add the pie chart to the list
plot_list[[column_name]] <- pie_chart
}
# Arrange the plots into a grid
plotgrid <- grid.arrange(grobs = plot_list, ncol = 4, returnGrob= TRUE)
Fichiers à télécharger :
Pour télécharger, cliquez avec le bouton droit de la souris et appuyez sur “Enregistrer le fichier sous” ou “Télécharger le fichier lié”.