P01 : Laboratoire de perception et d’attention
Sevda Montakhaby Nodeh
Laboratoire de perception et d’attention
En tant que chercheur en sciences cognitives au Cognition and Attention Lab de l'Université McMaster, vous êtes à la pointe de l'exploration des subtilités du contrôle proactif dans les processus d'attention. Cette ligne de recherche est d'une grande importance, étant donné que le système sensoriel humain est submergé par une vaste gamme d'informations à chaque seconde, dépassant ce qui peut être traité de manière significative. L'essence de la recherche sur l'attention consiste à décrypter les mécanismes par lesquels l'entrée sensorielle est parcourue et gérée de manière sélective. Cela est particulièrement important pour comprendre comment les individus anticipent et s'adaptent en vue de tâches ou de stimuli à venir, un phénomène particulièrement pertinent dans des environnements regorgeant de distractions potentielles.
Dans la vie de tous les jours, les conflits attentionnels sont monnaie courante et se manifestent lorsque des informations non pertinentes par rapport à un objectif entrent en compétition avec des données pertinentes par rapport à un objectif pour la priorité attentionnelle. Un exemple de ce phénomène (que nous ne connaissons que trop bien, j'en suis sûr) est la perturbation causée par les notifications sur nos appareils mobiles, qui peuvent nous détourner de nos objectifs principaux, tels que les études ou la conduite. D'un point de vue scientifique, l'élucidation des stratégies employées par le système cognitif humain pour optimiser la sélection et le maintien d'un comportement orienté vers un objectif représente un défi formidable et irrésistible.
Votre recherche en cours est une réponse directe à ce défi. Elle examine comment le contrôle proactif influence la capacité à se concentrer sur les informations pertinentes pour la tâche tout en écartant efficacement les distractions. Cette facette de la fonctionnalité cognitive n'est pas seulement une construction théorique ; c'est le fondement même du comportement et de l'interaction humaine au quotidien.
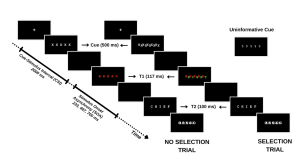
Votre étude évalue méthodiquement cette dynamique en engageant les participants dans une tâche où ils doivent identifier une séquence de mots dans des conditions variables. Le premier mot (T1) est présenté en rouge, suivi rapidement par un second mot (T2) en blanc. L'intervalle entre l'apparition de T1 et T2, connu sous le nom d'asynchronisme d'apparition du stimulus (SOA), sert de mesure critique dans votre expérience. La particularité de votre étude réside dans la façon dont vous manipulez les demandes d'attention sélective dans chaque essai, classées comme suit :
- Essais sans sélection : T1 apparaît seul, ce qui conduit généralement à une précision d'identification supérieure pour T1 et T2 en raison d'une charge cognitive réduite.
- Essais de sélection : T1 est entrecoupée d'un mot distracteur vert. Dans ces essais plus exigeants, le distracteur vert est en concurrence avec le mot cible rouge, ce qui entraîne une baisse de la précision d'identification pour T1 et T2.
En introduisant des indices informatifs et non informatifs, votre enquête sonde le rôle du contrôle proactif. Les indices informatifs donnent aux participants un aperçu du type d'épreuve à venir, ce qui leur permet de se préparer mentalement au défi imminent. À l'inverse, les indices non informatifs agissent comme des essais de contrôle et n'offrent aucune indication sur le type d'essai. L'hypothèse est que ces indices informatifs permettent aux participants d'ajuster leur attention de manière proactive en prévision d'un conflit attentionnel, ce qui pourrait améliorer les performances lors des essais de sélection avec des indices informatifs par rapport à ceux avec des indices non informatifs.
Pour une vue d'ensemble des différents types d'essais, veuillez vous référer à la figure ci-dessous. (le texte de la figure demeure toutefois en anglais seulement)
Dans le cadre de cette étude, vous vous attaquez non seulement à la question plus générale du rôle de l'effort conscient dans l'attention, mais vous contribuez également à une compréhension nuancée des processus cognitifs humains, ouvrant la voie à des applications qui vont de l'amélioration de la productivité de la vie quotidienne à l'optimisation des interfaces technologiques en vue d'une perturbation minimale de la cognition.
Mise en route : Chargement des progiciels , définition du répertoire de travail et chargement du jeu de données
Commençons par exécuter le code suivant dans RStudio pour charger les bibliothèques requises. Veillez à lire les commentaires intégrés dans le code pour comprendre ce que fait chaque ligne de code.
Remarque : les cases grisées contiennent le code R, le signe "#" indiquant un commentaire qui ne s'exécutera pas dans RStudio.
# Here we create a list called "my_packages" with all of our required libraries
my_packages <- c("tidyverse", "rstatix", "readxl", "xlsx", "emmeans", "afex",
"kableExtra", "grid", "gridExtra", "superb", "ggpubr", "lsmeans")
# Checking and extracting packages that are not already installed
not_installed <- my_packages[!(my_packages %in% installed.packages()[ , "Package"])]
# Install packages that are not already installed
if(length(not_installed)) install.packages(not_installed)
# Loading the required libraries
library(tidyverse) # for data manipulation
library(rstatix) # for statistical analyses
library(readxl) # to read excel files
library(xlsx) # to create excel files
library(kableExtra) # formatting html ANOVA tables
library(superb) # production of summary stat with adjusted error bars(Cousineau, Goulet, & Harding, 2021)
library(ggpubr) # for making plots
library(grid) # for plots
library(gridExtra) # for arranging multiple ggplots for extraction
library(lsmeans) # for pairwise comparisons
Assurez-vous d'avoir téléchargé l'ensemble de données requis ("ProactiveControlCueing.xlsx") pour cet exercice. Définissez le répertoire de travail de votre session R actuelle dans le dossier contenant l'ensemble de données téléchargé. Vous pouvez le faire manuellement dans le studio R en cliquant sur l'onglet "Session" en haut de l'écran, puis en cliquant sur "Set Working Directory".
Si le fichier de données téléchargé et votre session R se trouvent dans le même dossier, vous pouvez choisir de définir votre répertoire de travail sur "l'emplacement du fichier source" (l'emplacement où votre session R actuelle est sauvegardée). S'ils se trouvent dans des dossiers différents, cliquez sur l'option "choisir un répertoire" et recherchez l'emplacement du jeu de données téléchargé.
Vous pouvez également effectuer cette opération en exécutant le code suivant
setwd(file.choose())
Une fois que vous avez défini votre répertoire de travail, manuellement ou par code, la console ci-dessous affiche le répertoire complet de votre dossier.
Lisez l'ensemble de données téléchargé sous le nom de "cueingData" et effectuez les exercices qui l'accompagnent au mieux de vos capacités.
# Read xlsx file
cueingData = read_excel("ProactiveControlCueing.xlsx")
Fichiers à télécharger :
Solutions
Exercice 1 - Préparation et exploration des données
Après avoir configuré les progiciels nécessaires, établi votre répertoire de travail et chargé l'ensemble de données ("cueingData") dans RStudio, procédez aux exercices ci-dessous. Copiez et collez votre code R dans les zones de texte prévues à cet effet. Vous pouvez exporter les exercices et vos réponses à la fin de cet exercice en tant que fichier docx à partir de la page d'exportation de documents une fois que vous avez terminé.
Une fois les exercices terminés, comparez vos solutions au corrigé inclus ci-dessous. N'oubliez pas que RStudio peut produire des résultats identiques par le biais de différentes méthodes. Ne vous découragez donc pas si votre code diffère du corrigé, à condition que vos résultats soient corrects.
- Affichez les premières rangées de vos données
head(cueingData) #Displaying the first few rows## # A tibble: 6 × 6
## ID CUE_TYPE TRIAL_TYPE SOA T1Score T2Score
## <dbl> <chr> <chr> <dbl> <dbl> <dbl>
## 1 1 INFORMATIVE NS 233 100 94.6
## 2 2 INFORMATIVE NS 233 100 97.2
## 3 3 INFORMATIVE NS 233 89.2 93.9
## 4 4 INFORMATIVE NS 233 100 91.9
## 5 5 INFORMATIVE NS 233 100 100
## 6 6 INFORMATIVE NS 233 100 97.3
2. Définissez vos facteurs et vérifiez leur structure. Assurez-vous que vos mesures dépendantes sont sous forme numérique et que vos facteurs et niveaux sont correctement configurés.
cueingData <- cueingData %>%
convert_as_factor(ID, CUE_TYPE, TRIAL_TYPE, SOA) #setting up factors
str(cueingData) #checking that factors and levels are set-up correctly. Checking to see that dependent measures are in numerical format.
## # A tibble: 6 × 6
## ID CUE_TYPE TRIAL_TYPE SOA T1Score T2Score
## <dbl> <chr> <chr> <dbl> <dbl> <dbl>
## 1 1 INFORMATIVE NS 233 100 94.6
## 2 2 INFORMATIVE NS 233 100 97.2
## 3 3 INFORMATIVE NS 233 89.2 93.9
## 4 4 INFORMATIVE NS 233 100 91.9
## 5 5 INFORMATIVE NS 233 100 100
## 6 6 INFORMATIVE NS 233 100 97.3
## tibble [192 × 6] (S3: tbl_df/tbl/data.frame)
## $ ID : Factor w/ 16 levels "1","2","3","4",..: 1 2 3 4 5 6 7 8 9 10 ...
## $ CUE_TYPE : Factor w/ 2 levels "INFORMATIVE",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ TRIAL_TYPE: Factor w/ 2 levels "NS","S": 1 1 1 1 1 1 1 1 1 1 ...
## $ SOA : Factor w/ 3 levels "233","467","700": 1 1 1 1 1 1 1 1 1 1 ...
## $ T1Score : num [1:192] 100 100 89.2 100 100 ...
## $ T2Score : num [1:192] 94.6 97.2 93.9 91.9 100 ...
3. Effectuer des contrôles de base des données pour vérifier les valeurs manquantes et la cohérence des données
sum(is.na(cueingData)) # Checking for missing values in the dataset
## [1] 0
summary(cueingData) # Viewing the summary of the dataset to check for inconsistencies
## ID CUE_TYPE TRIAL_TYPE SOA T1Score
## 1 : 12 INFORMATIVE :96 NS:96 233:64 Min. : 32.43
## 2 : 12 UNINFORMATIVE:96 S :96 467:64 1st Qu.: 77.78
## 3 : 12 700:64 Median : 95.87
## 4 : 12 Mean : 86.33
## 5 : 12 3rd Qu.:100.00
## 6 : 12 Max. :100.00
## (Other):120
## T2Score
## Min. : 29.63
## 1st Qu.: 83.97
## Median : 95.76
## Mean : 87.84
## 3rd Qu.:100.00
## Max. :100.00
- Vos données correspondent-elles à un plan équilibré ou déséquilibré ? (Conseil : utilisez un code pour indiquer le nombre d'observations par combinaison de facteurs)
table(cueingData$CUE_TYPE, cueingData$TRIAL_TYPE, cueingData$SOA) #checking the number of observations per condition or combination of factors. Data is a balanced design since there is an equal number of observations per cell.
## , , = 233
##
##
## NS S
## INFORMATIVE 16 16
## UNINFORMATIVE 16 16
##
## , , = 467
##
##
## NS S
## INFORMATIVE 16 16
## UNINFORMATIVE 16 16
##
## , , = 700
##
##
## NS S
## INFORMATIVE 16 16
## UNINFORMATIVE 16 16
Exercice 2 - Calculer les statistiques sommaires
Nous utiliserons ici la bibliothèque Superb pour calculer nos statistiques sommaires avec l'erreur standard des mesures moyennes qui ont été corrigées pour les comparaisons entre sujets.
Pour vous familiariser avec la bibliothèque Superb, je vous suggère de lire l'article publié suivant et de regarder les tutoriels YouTube.
Cousineau D, Goulet M, Harding B (2021). "Graphiques sommaires avec barres d'erreur ajustées : The superb framework with an implementation in R." Advances in Methods and Practices in Psychological Science, 2021, 1-46. doi : https://doi.org/10.1177/25152459211035109
Walker, J. A. L. (2021). "Summary plots with adjusted error bars (superb)". Vidéo Youtube, accessible ici.
Walker, J. A. L. (2021). Summary plots with adjusted error bars (superb). Extrait de https://www.youtube.com/watch?v=rw_6ll5nVus
Pour jouer avec les différentes fonctionnalités de la bibliothèque superb, une application Shiny avec une vue de constructeur pour la bibliothèque est également disponible sur le web. Vous trouverez également ci-dessous une ressource utile pour naviguer dans le code R de la bibliothèque Superb.
5. La bibliothèque Superb exige que votre jeu de données soit dans un format large. Convertissez donc votre jeu de données d'un format long à un format large. Enregistrez-le sous "cueingData.wide".
cueingData.wide <- cueingData %>%
pivot_wider(names_from = c(TRIAL_TYPE, SOA, CUE_TYPE),
values_from = c(T1Score, T2Score) )
6. En utilisant superbPlot() et cueingData.wide, calculez la moyenne et l'erreur standard de la moyenne (SEM) pour les scores T1 et T2 à chaque niveau des facteurs. Veillez à calculer les valeurs SEM corrigées de Cousineau-Morey.
- Vous devez le faire séparément pour chacune de vos mesures dépendantes. Enregistrez votre fonction superbplot pour T1Score sous "EXP1.T1.plot" et sous "EXP1.T2.plot" pour T2Score.
- Renommez les niveaux des facteurs dans chaque graphique. Actuellement, les niveaux sont numérotés. Nous voulons que les niveaux de SOA soient 233, 467 et 700 ; que les niveaux de type de repère soient Informatif et Non informatif, et que les niveaux de type d'essai soient Sélection et Pas de sélection (Astuce : pour accéder aux données récapitulatives, utilisez EXP1.T1.plot$data$insertfactorname).
EXP1.T1.plot <- superbPlot(cueingData.wide,
WSFactors = c("SOA(3)", "CueType(2)", "TrialType(2)"),
variables = c("T1Score_NS_233_INFORMATIVE", "T1Score_NS_467_INFORMATIVE",
"T1Score_NS_700_INFORMATIVE", "T1Score_NS_233_UNINFORMATIVE",
"T1Score_NS_467_UNINFORMATIVE", "T1Score_NS_700_UNINFORMATIVE",
"T1Score_S_233_INFORMATIVE", "T1Score_S_467_INFORMATIVE",
"T1Score_S_700_INFORMATIVE", "T1Score_S_233_UNINFORMATIVE",
"T1Score_S_467_UNINFORMATIVE", "T1Score_S_700_UNINFORMATIVE"),
statistic = "mean",
errorbar = "SE",
adjustments = list(
purpose = "difference",
decorrelation = "CM",
popSize = 32
),
plotStyle = "line",
factorOrder = c("SOA", "CueType", "TrialType"),
lineParams = list(size=1, linetype="dashed"),
pointParams = list(size = 3))
## superb::FYI: Here is how the within-subject variables are understood:
## SOA CueType TrialType variable
## 1 1 1 T1Score_NS_233_INFORMATIVE
## 2 1 1 T1Score_NS_467_INFORMATIVE
## 3 1 1 T1Score_NS_700_INFORMATIVE
## 1 2 1 T1Score_NS_233_UNINFORMATIVE
## 2 2 1 T1Score_NS_467_UNINFORMATIVE
## 3 2 1 T1Score_NS_700_UNINFORMATIVE
## 1 1 2 T1Score_S_233_INFORMATIVE
## 2 1 2 T1Score_S_467_INFORMATIVE
## 3 1 2 T1Score_S_700_INFORMATIVE
## 1 2 2 T1Score_S_233_UNINFORMATIVE
## 2 2 2 T1Score_S_467_UNINFORMATIVE
## 3 2 2 T1Score_S_700_UNINFORMATIVE
## superb::FYI: The HyunhFeldtEpsilon measure of sphericity per group are 0.134
## superb::FYI: Some of the groups' data are not spherical. Use error bars with caution.
EXP1.T2.plot <- superbPlot(cueingData.wide,
WSFactors = c("SOA(3)", "CueType(2)", "TrialType(2)"),
variables = c("T2Score_NS_233_INFORMATIVE", "T2Score_NS_467_INFORMATIVE",
"T2Score_NS_700_INFORMATIVE", "T2Score_NS_233_UNINFORMATIVE",
"T2Score_NS_467_UNINFORMATIVE", "T2Score_NS_700_UNINFORMATIVE",
"T2Score_S_233_INFORMATIVE", "T2Score_S_467_INFORMATIVE",
"T2Score_S_700_INFORMATIVE", "T2Score_S_233_UNINFORMATIVE",
"T2Score_S_467_UNINFORMATIVE", "T2Score_S_700_UNINFORMATIVE"),
statistic = "mean",
errorbar = "SE",
adjustments = list(
purpose = "difference",
decorrelation = "CM",
popSize = 32
),
plotStyle = "line",
factorOrder = c("SOA", "CueType", "TrialType"),
lineParams = list(size=1, linetype="dashed"),
pointParams = list(size = 3)
)
## superb::FYI: Here is how the within-subject variables are understood:
## SOA CueType TrialType variable
## 1 1 1 T2Score_NS_233_INFORMATIVE
## 2 1 1 T2Score_NS_467_INFORMATIVE
## 3 1 1 T2Score_NS_700_INFORMATIVE
## 1 2 1 T2Score_NS_233_UNINFORMATIVE
## 2 2 1 T2Score_NS_467_UNINFORMATIVE
## 3 2 1 T2Score_NS_700_UNINFORMATIVE
## 1 1 2 T2Score_S_233_INFORMATIVE
## 2 1 2 T2Score_S_467_INFORMATIVE
## 3 1 2 T2Score_S_700_INFORMATIVE
## 1 2 2 T2Score_S_233_UNINFORMATIVE
## 2 2 2 T2Score_S_467_UNINFORMATIVE
## 3 2 2 T2Score_S_700_UNINFORMATIVE
## superb::FYI: The HyunhFeldtEpsilon measure of sphericity per group are 0.226
## superb::FYI: Some of the groups' data are not spherical. Use error bars with caution.
# Re-naming levels of the factors
levels(EXP1.T1.plot$data$SOA) <- c("1" = "233", "2" = "467", "3" = "700")
levels(EXP1.T2.plot$data$SOA) <- c("1" = "233", "2" = "467", "3" = "700")
levels(EXP1.T1.plot$data$TrialType) <- c("1" = "No Selection", "2" = "Selection")
levels(EXP1.T2.plot$data$TrialType) <- c("1" = "No Selection", "2" = "Selection")
levels(EXP1.T1.plot$data$CueType) <- c("1" = "Informative", "2" = "Uninformative")
levels(EXP1.T2.plot$data$CueType) <- c("1" = "Informative", "2" = "Uninformative")
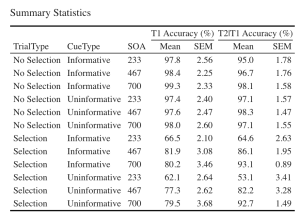
7. Créons un magnifique tableau HTML imprimable des statistiques récapitulatives pour les scores T1 et T2. Ce tableau récapitulatif peut ensuite être utilisé dans votre manuscrit. Je vous suggère de visiter le lien suivant pour obtenir des guides sur la façon de créer des tableaux imprimables. Personnalisation du tableau HTML
- Commencez par extraire les données de statistiques sommaires avec les moyennes de groupe et les valeurs SEM de CousineauMorey de chaque fonction de tracé et enregistrez-les en tant que cadre de données séparément pour les données T1 et T2 (vous devriez avoir deux cadres de données avec vos statistiques sommaires nommés "EXP1.T1.summaryData" et "EXP1.T2.summaryData").
- Dans vos deux cadres de données avec les statistiques sommaires, arrondissez vos moyennes à une décimale et vos valeurs SEM à deux décimales.
- Fusionnez les données récapitulatives de T1Score et T2Score et enregistrez-les sous "EXP1_summarystat_results"
- Dans ce tableau fusionné, supprimez les colonnes contenant les valeurs SEM négatives (valeurs SEM de largeur inférieure).
- Renommez les colonnes de ce cadre de données fusionné de sorte que le nom des colonnes contenant les moyennes T1Score et T2Score soit "Means" et que les colonnes contenant les scores SEM pour l'une ou l'autre des variables dépendantes soient "SEM".
- Intitulez votre tableau "Statistiques sommaires"
- Réglez la police de votre texte sur "Cambria" et la taille de la police sur 14.
- Définissez les en-têtes des colonnes T1Score means et SEM comme "T1 Accuracy (%)".
- Définissez les en-têtes des colonnes T2Score means et SEM comme "T2 Accuracy (%)".
# Extracting summary data with CousineauMorey SEM Bars
EXP1.T1.summaryData <- data.frame(EXP1.T1.plot$data)
EXP1.T2.summaryData <- data.frame(EXP1.T2.plot$data)
# Rounding values in each column
# round(x, 1) rounds to the specified number of decimal places
EXP1.T1.summaryData$center <- round(EXP1.T1.summaryData$center,1)
EXP1.T1.summaryData$upperwidth <- round(EXP1.T1.summaryData$upperwidth,2)
EXP1.T2.summaryData$center <- round(EXP1.T2.summaryData$center,1)
EXP1.T2.summaryData$upperwidth <- round(EXP1.T2.summaryData$upperwidth,2)
# merging T1 and T2|T1 summary tables
EXP1_summarystat_results <- merge(EXP1.T1.summaryData, EXP1.T2.summaryData, by=c("TrialType","CueType","SOA"))
# Rename the column name
colnames(EXP1_summarystat_results)[colnames(EXP1_summarystat_results) == "center.x"] ="Mean"
colnames(EXP1_summarystat_results)[colnames(EXP1_summarystat_results) == "center.y"] ="Mean"
colnames(EXP1_summarystat_results)[colnames(EXP1_summarystat_results) == "upperwidth.x"] ="SEM"
colnames(EXP1_summarystat_results)[colnames(EXP1_summarystat_results) == "upperwidth.y"] ="SEM"
# deleting columns by name "lowerwidth.x" and "lowerwidth.y" in each summary table
EXP1_summarystat_results <- EXP1_summarystat_results[ , ! names(EXP1_summarystat_results) %in% c("lowerwidth.x", "lowerwidth.y")]
#removing suffixes from column names
colnames(EXP1_summarystat_results)<-gsub(".1","",colnames(EXP1_summarystat_results))
# Printable ANOVA html
EXP1_summarystat_results %>%
kbl(caption = "Summary Statistics") %>%
kable_classic(full_width = F, html_font = "Cambria", font_size = 14) %>%
add_header_above(c(" " = 3, "T1 Accuracy (%)" = 2, "T2|T1 Accuracy (%)" = 2))
Exercice 3 - Visualiser les données
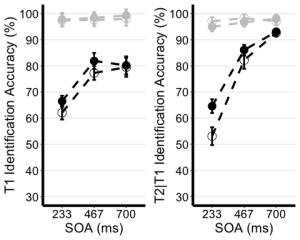
8. Utilisez le tableau de statistiques récapitulatives non édité de l'exercice 2 (EXP1.T1.summaryData et EXP1.T3.summaryData) et la fonction ggplot() pour créer des graphiques linéaires récapitulatifs distincts pour les scores T1 et T2. Le graphique linéaire visualise la relation entre la SOA et la mesure dépendante tout en tenant compte des facteurs de type de repère et de type d'essai. Votre graphique doit présenter les caractéristiques suivantes :
- Tracer le SOA sur l'axe des abscisses et l'intituler "SOA (ms)"
- Tracez la mesure dépendante sur l'axe des ordonnées et intitulez l'axe des ordonnées "Précision d'identification T1" pour le tracé T1, et "Précision d'identification T2|T1 (%)" pour le tracé T2.
- Définissez la couleur de vos lignes en fonction du type d'essai.
- Définissez la forme des points pour chaque valeur de la ligne par type d'indice.
- Utilisez la fonction geom_point() pour personnaliser la forme de vos points. Utilisez des cercles pleins pour les indices informatifs et des cercles creux pour les indices non informatifs.
- Utilisez la fonction scale_color_manual() pour personnaliser les couleurs des lignes. Réglez la couleur des lignes représentant les essais de sélection sur "black" et celle des lignes représentant les essais de non-sélection sur "gray78".
- Utilisez geom_line() pour personnaliser le type de ligne. Réglez le type de ligne sur pointillé et la largeur de ligne sur 1,2.
- Personnalisez votre axe des y. Fixez la valeur minimale à 30 et la valeur maximale à 100.
- Utilisez la fonction scale_y_continuous() pour que les valeurs de l'axe des y augmentent par incréments de 10.
- Utilisez la fonction geom_errorbar() pour tracer des barres d'erreur en utilisant les valeurs SEM calculées dans le tableau récapitulatif.
- Définissez le thème du tracé sur theme_classic()
- Utilisez la fonction theme() pour personnaliser la taille de la police de l'axe des x et la largeur des lignes. Modifier la taille de la police du titre principal de l'axe à 16, et les étiquettes de l'axe des x à 14.
- Ajoutez des lignes de grille horizontales.
- Ne pas inclure de légende.
- Stockez vos deux graphiques sous les noms "T1.ggplotplot" et "T2.ggplotplot"
EXP1.T1.ggplotplot <- ggplot(EXP1.T1.summaryData, aes(x=SOA, y=center, color=TrialType, shape = CueType,
group=interaction(CueType, TrialType))) +
geom_point(data=filter(EXP1.T1.summaryData, CueType == "Uninformative"), shape=1, size=4.5) + # assigning shape type to level of factor
geom_point(data=filter(EXP1.T1.summaryData, CueType == "Informative"), shape=16, size=4.5) + # assigning shape type to level of factor
geom_line(linetype="dashed", linewidth=1.2) + # change line thickness and line style
scale_color_manual(values = c("gray78", "black") ) +
xlab("SOA (ms)") +
ylab("T1 Identification Accuracy (%)") +
theme_classic() + # It has no background, no bounding box.
theme(axis.line=element_line(size=1.5), # We make the axes thicker...
axis.text = element_text(size = 14, colour = "black"), # their text bigger...
axis.title = element_text(size = 16, colour = "black"), # their labels bigger...
panel.grid.major.y = element_line(), # adding horizontal grid lines
legend.position = "none") +
coord_cartesian(ylim=c(30, 100)) +
scale_y_continuous(breaks=seq(30, 100, 10)) + # Ticks from 30-100, every 10
geom_errorbar(aes(ymin=center+lowerwidth, ymax=center+upperwidth), width = 0.12, size = 1) # adding error bars from summary table
EXP1.T2.ggplotplot <- ggplot(EXP1.T2.summaryData, aes(x=SOA, y=center, color=TrialType, shape=CueType,
group=interaction(CueType, TrialType))) +
geom_point(data=filter(EXP1.T2.summaryData, CueType == "Uninformative"), shape=1, size=4.5) + # assigning shape type to level of factor
geom_point(data=filter(EXP1.T2.summaryData, CueType == "Informative"), shape=16, size=4.5) + # assigning shape type to level of factor
geom_line(linetype="dashed", linewidth=1.2) + # change line thickness and line style
scale_color_manual(values = c("gray78", "black")) +
xlab("SOA (ms)") +
ylab("T2|T1 Identification Accuracy (%)") +
theme_classic() + # It has no background, no bounding box.
theme(axis.line=element_line(size=1.5), # We make the axes thicker...
axis.text = element_text(size = 14, colour = "black"), # their text bigger...
axis.title = element_text(size = 16, colour = "black"), # their labels bigger...
panel.grid.major.y = element_line(), # adding horizontal grid lines
legend.position = "none") +
guides(fill = guide_legend(override.aes = list(shape = 16) ),
shape = guide_legend(override.aes = list(fill = "black"))) +
coord_cartesian(ylim=c(30, 100)) +
scale_y_continuous(breaks=seq(30, 100, 10)) + # Ticks from 30-100, every 10
geom_errorbar(aes(ymin=center+lowerwidth, ymax=center+upperwidth), width = 0.12, size = 1) # adding error bars from summary table
9. Utilisez ggarrange() pour afficher vos tracés ensemble.
ggarrange(EXP1.T1.ggplotplot, EXP1.T2.ggplotplot,
nrow = 1, ncol = 2, common.legend = F,
widths = 8, heights = 5)
Exercice 4 - L’analyse principale
Étant donné que les performances des essais de non-sélection sont proches du plafond (près de 100 %), nous concentrerons nos analyses sur les essais de sélection.
10. Utilisez les données en format long ("cueingData") et la fonction anova_test(), et calculez une ANOVA à deux voies pour chaque variable dépendante, mais sur les essais de sélection uniquement. Définissez le type de repère et l'AOS comme facteurs internes aux participants. (Conseil : utilisez la fonction filter())
- Réglez votre mesure de l'ampleur de l'effet sur l'éta quadratique partiel (pes).
- Assurez-vous de générer le tableau ANOVA détaillé.
- Enregistrez vos calculs et "T1_2anova" et "T2_2anova".
- Utilisez la fonction get_anova_table() pour afficher vos tableaux d'ANOVA.
T1_2anova <- anova_test(
data = filter(cueingData, TRIAL_TYPE == "S"), dv = T1Score, wid = ID,
within = c(CUE_TYPE, SOA), detailed = TRUE, effect.size = "pes")
T2_2anova <- anova_test(
data = filter(cueingData, TRIAL_TYPE == "S"), dv = T2Score, wid = ID,
within = c(CUE_TYPE, SOA), detailed = TRUE, effect.size = "pes")
get_anova_table(T1_2anova)
## ANOVA Table (type III tests)
##
## Effect DFn DFd SSn SSd F p p<.05 pes
## 1 (Intercept) 1.00 15.00 534043.211 30705.523 260.886 6.80e-11 * 0.946
## 2 CUE_TYPE 1.00 15.00 253.665 506.454 7.513 1.50e-02 * 0.334
## 3 SOA 1.29 19.35 5091.853 2660.275 28.710 1.16e-05 * 0.657
## 4 CUE_TYPE:SOA 2.00 30.00 77.140 1061.051 1.091 3.49e-01 0.068
get_anova_table(T2_2anova)
## ANOVA Table (type III tests)
##
## Effect DFn DFd SSn SSd F p p<.05 pes
## 1 (Intercept) 1 15 593913.106 11288.706 789.169 2.19e-14 * 0.981
## 2 CUE_TYPE 1 15 661.761 426.947 23.250 2.24e-04 * 0.608
## 3 SOA 2 30 20001.563 3852.629 77.875 1.33e-12 * 0.838
## 4 CUE_TYPE:SOA 2 30 519.154 1298.144 5.999 6.00e-03 * 0.286
Exercice 5 - Les tests post-hoc
S'il existe une interaction bidirectionnelle significative, cela signifie que l'impact d'un facteur est influencé par un autre facteur. Cela signifie que les deux variables indépendantes interagissent l'une avec l'autre pour produire un effet significatif sur la variable dépendante. Étant donné qu'il existe une interaction bidirectionnelle significative entre le type de repère et l'AOS pour les scores T2, nous devons procéder à des tests post hoc afin d'explorer et de comprendre les différences spécifiques entre les niveaux ou les conditions des facteurs impliqués dans l'interaction. Cela peut nous aider à identifier les combinaisons spécifiques à l'origine de l'effet d'interaction.
11. Filtrez d'abord les essais de sélection, puis regroupez vos données par SOA et utilisez la fonction pairwise_t_test() pour comparer les essais informatifs et non informatifs à chaque niveau de SOA, stockez et affichez votre calcul sous la forme "T2_sel_pwc".
T2_sel_pwc <- filter(cueingData, TRIAL_TYPE == "S") %>%
group_by(SOA) %>%
pairwise_t_test(T2Score ~ CUE_TYPE, paired = TRUE, p.adjust.method = "holm", detailed = TRUE) %>%
add_significance("p.adj")
T2_sel_pwc <- get_anova_table(T2_sel_pwc)
T2_sel_pwc
## # A tibble: 3 × 16
## SOA estimate .y. group1 group2 n1 n2 statistic p df
## <fct> <dbl> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl>
## 1 233 11.5 T2Score INFORMATIVE UNINFO… 16 16 4.36 5.55e-4 15
## 2 467 3.89 T2Score INFORMATIVE UNINFO… 16 16 1.97 6.8 e-2 15
## 3 700 0.356 T2Score INFORMATIVE UNINFO… 16 16 0.190 8.52e-1 15
## # ℹ 6 more variables: conf.low <dbl>, conf.high <dbl>, method <chr>,
## # alternative <chr>, p.adj <dbl>, p.adj.signif <chr>
Remarques finales
Superbe travail !
Vous avez réussi à reconstruire une figure et à effectuer des analyses dans le cadre d'un projet de mémoire de maîtrise. Les données sur lesquelles vous avez travaillé ont été recueillies dans le laboratoire d'attention et de mémoire du département de psychologie, de neuroscience et de comportement (PNB) de l'Université McMaster. Ce laboratoire prospère sous l'égide du Dr Bruce Milliken, dont les recherches sont profondément ancrées dans le domaine de la cognition humaine. L'objectif principal du laboratoire est d'élucider les processus fondamentaux qui sous-tendent l'attention, la mémoire et le contrôle cognitif. L'étendue des recherches menées ici englobe un large éventail de sujets. Il s'agit notamment d'étudier les nuances qui différencient les processus mentaux conscients et inconscients, d'explorer la manière dont les mécanismes attentionnels influencent et soutiennent l'apprentissage et la mémoire, de comprendre le rôle de l'imagerie visuelle dans la formation de l'attention et de la perception, et d'examiner l'impact de l'apprentissage implicite sur l'orchestration du contrôle de l'attention. Chaque projet au sein de ce laboratoire témoigne de l'engagement à faire progresser notre compréhension de la tapisserie complexe de la cognition humaine.
Références et lectures complémentaires :
Si vous souhaitez approfondir l'ensemble des données et les spécificités du projet de thèse, je vous encourage à explorer la thèse de doctorat librement accessible ou l'article publié cité ci-dessous :
Montakhaby Nodeh, S., MacLellan, E., & Milliken, B. (2024). Proactive control: Endogenous cueing effects in a two-target attentional blink task. Consciousness and Cognition, 118, 103648. Elsevier BV. https://doi.org/10.1016/j.concog.2024.103648

