1
1
Contexte
Les protéines jouent un rôle essentiel dans la plupart des processus biologiques, notamment en tant que catalyseurs de réactions physiologiques, régulateurs de ces réactions ou cadre structurel autour duquel ces processus peuvent se produire. L’organisation complexe des diverses fonctionnalités des protéines dans l’espace 3D permet aux organismes vivants de disposer d’une surprenante gamme de fonctions. La compréhension de cette relation intime entre la structure et la fonction est au cœur de la compréhension du monde naturel et la clé de son contrôle.
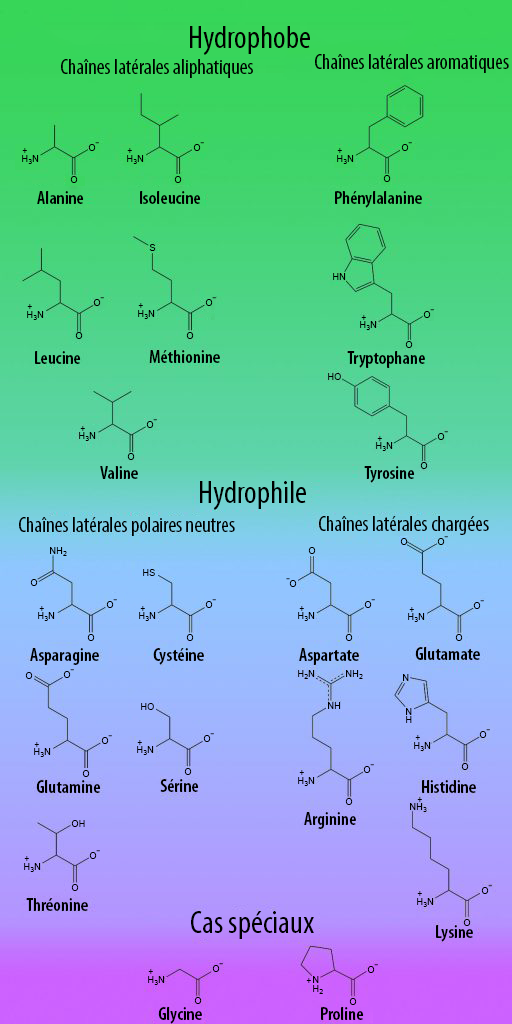
La mutagenèse dirigée (le remplacement d’un codon d’acide aminé par un autre dans un gène) est un outil courant et pratique pour étudier la fonction des protéines. Cependant, malgré leur complexité fonctionnelle, les protéines ont une base structurelle assez simple : tous les organismes utilisent les mêmes 20 acides aminés pour construire les protéines, qui ont toutes une fonctionnalité chimique limitée (figure 1). Par conséquent, afin d’élargir la gamme d’études pouvant être réalisées sur les protéines, les acides aminés non canoniques (ou ncAA, aussi appelés acides aminés non naturels ou UAA), dont la fonctionnalité chimique est illimitée, sont des outils évidents pour étudier ces relations entre la structure et la fonction.
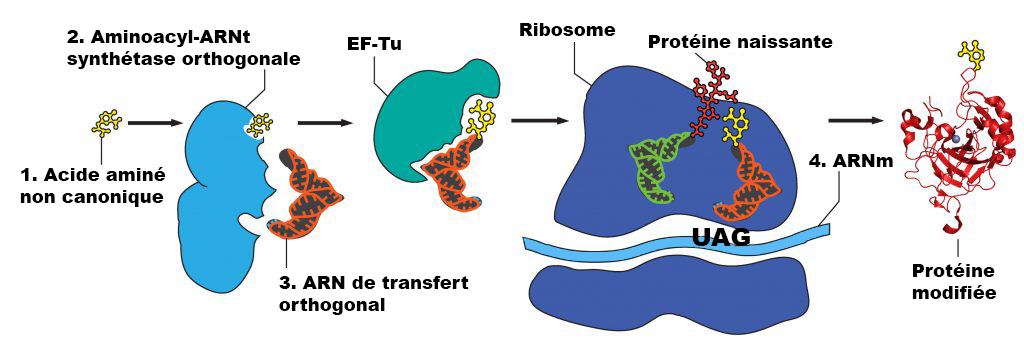
La manipulation des protéines pour permettre l’utilisation des ncAA exige l’exploitation du dogme central de la biologie moléculaire : la transcription d’une séquence d’ADN en ARNm et la traduction de cette séquence d’ARNm en une chaîne peptidique repliable et fonctionnelle. Un changement à l’ADN causera donc un changement dans la protéine. Cependant, la nature du code génétique, comportant plusieurs codons dégénérés spécifiant le même acide aminé, ne laisse pas de codons « inutilisés » permettant d’ajouter facilement des ncAA in vivo. Le codon doit donc être « détourné » pour pouvoir spécifier le ncAA en remplaçant un codon naturel dans une séquence. Le codon TAG, le moins utilisé des trois codons STOP, joue ce rôle et peut facilement remplacer n’importe quel codon existant dans une séquence grâce à la mutagenèse dirigée. Afin d’empêcher ce codon de signaler la fin de la chaîne peptidique, il doit fonctionner comme les autres codons « naturels » : une aminoacyl-ARNt synthétase (RS) doit lier un acide aminé à un ARNt personnalisé, qui ajoute ensuite l’acide aminé à la chaîne peptidique par l’intermédiaire du ribosome (figure 2). Étant donné que l’acide aminé n’est pas produit naturellement, il n’y a pas de paire RS/ARNt naturellement présente dans la cellule.
Pour faciliter l’incorporation spécifique du ncAA, la cellule doit donc disposer d’un ncAA-RS qui permet l’aminoacylation de l’ARNt couplé avec le ncAA uniquement, mais pas avec les acides aminés endogènes. Pour cela, il faut d’abord trouver dans la nature une RS et un ARNt utilisables par une cellule hôte (orthogonaux). Cette paire doit pouvoir fonctionner ensemble et avec le ribosome d’E. coli, et l’ARNt doit être capable de reconnaître le codon TAG de manière spécifique. Un nouveau ncAA non toxique peut être choisi pour les propriétés chimiques intéressantes qu’il peut introduire dans la protéine. Il faut ensuite sélectionner une RS capable de lier le ncAA pertinent lors de l’aminoacylation de l’ARNt. L’évolution dirigée des RS implique des cycles de sélection positive et négative sur une liste de RS (jusqu’à 108 membres), qui partagent toutes la même structure globale, mais présentent une variété de mutations concentrées autour du site de liaison de l’acide aminé. Les cycles de sélection positive consistent à transformer les membres de la liste en cellules contenant un plasmide avec le gène du chloramphénicol acétyl transférase (CAT), qui confère la résistance au chloramphénicol, contenant un codon TAG. Les cellules sont ensuite cultivées en présence du ncAA et de l’antibiotique chloramphénicol; les membres qui parviennent à incorporer le ncAA (ainsi que ceux qui incorporent des acides aminés endogènes) produisent une protéine CAT complètement fonctionnelle et peuvent survivre dans le milieu contenant du chloramphénicol. Pour exclure les membres restants qui ont utilisé un acide aminé endogène à la place du ncAA, un cycle de sélection négative est ensuite effectué. Les cycles de sélection négative consistent à isoler les plasmides contenant la RS des cellules ayant survécu à la sélection positive, puis à transformer les membres restants de la RS en cellules contenant un autre plasmide. Ce plasmide contient le gène toxique de la barnase avec un codon TAG en son milieu, qui encode une protéine toxique qui tue la cellule si elle est produite avec succès. Le ncAA est exclu du milieu dans les cycles de sélection négative, de sorte que les RS qui incorporent un acide aminé naturel en réponse au codon TAG ne survivent pas. Plusieurs cycles alternés de sélection positive et négative sont effectués jusqu’à ce que les RS restantes soient celles qui peuvent lier efficacement un ncAA à un ARNt (et donc produire une protéine contenant un codon TAG), mais qui ne peuvent pas lier un acide aminé naturel à cet ARNt.
Pour que la cellule produise la protéine TAG mutante et la paire ncAA-RS/ARNt nécessaire, il faut lui fournir les gènes qui les encodent. Les gènes sont introduits dans les cellules d’E. coli à l’aide des plasmides pBad et pDule (figure 3).
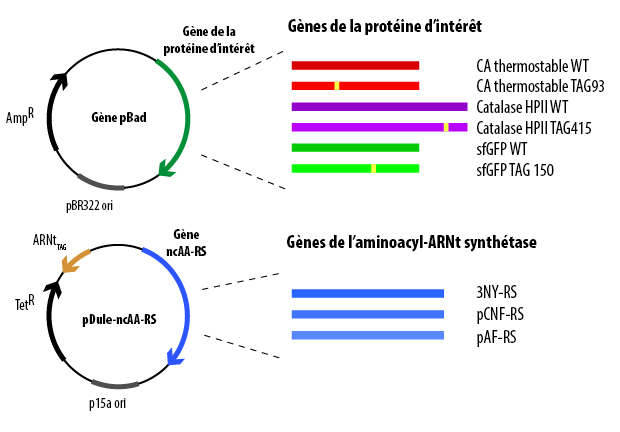
Le plasmide pBad contient le gène (lui-même contenant un codon TAG) qui encode la protéine pertinente contrôlée par un promoteur induit par l’arabinose, qui est une origine de la réplication, et un gène qui encode la bêta-lactamase (qui confère une résistance à l’ampicilline). Lorsqu’ils sont clonés dans le plasmide pBad, les codons pour 6 histidines sont ajoutés à la terminaison N ou C du gène, ce qui permet la purification par affinité de la protéine surexprimée. Le plasmide pDule contient les gènes qui encodent la ncAA-RS et l’ARNt(CUA), une origine de réplication (qui doit être compatible avec l’origine de réplication de pBAD) et un gène qui encode la protéine TetA (qui confère la résistance à la tétracycline). Ces plasmides doivent tous deux être transformés dans la cellule pour que la protéine de pleine longueur contenant le ncAA soit produite. Pour vérifier que les cellules contiennent les deux plasmides avant de commencer la surexpression, les cellules sont cultivées en présence des antibiotiques ampicilline et tétracycline, ce qui garantit que seules les cellules contenant les deux plasmides pourront se développer dans le milieu.
Les cellules contenant les plasmides adaptés peuvent ensuite être induites pour surexprimer la protéine pertinente à l’aide d’un milieu d’auto-induction à base d’arabinose. Le plasmide pBad contient un système promoteur d’arabinose qui active l’expression du gène sur ce plasmide en présence d’arabinose. Le milieu d’auto-induction est conçu pour permettre aux cellules d’atteindre une densité élevée avant que la surexpression soit induite, afin qu’un plus grand nombre de cellules soit disponible pour surexprimer la protéine lorsque l’induction commence. L’auto-induction du milieu est réalisée en utilisant des concentrations de sucre définies : lorsque les niveaux de glucose commencent à diminuer en raison du métabolisme cellulaire et de la croissance, les cellules commencent à absorber l’arabinose disponible. Bien qu’elles ne puissent pas le métaboliser pour poursuivre leur croissance, l’arabinose fonctionne comme un activateur du promoteur du plasmide pBad, induisant ainsi la surexpression de la protéine. L’électrophorèse sur gel des protéines brutes permet ensuite de vérifier facilement le succès de ce processus en contrôlant la taille de la protéine (de pleine longueur ou tronquée) produite en présence ou en l’absence de ncAA. La production d’une protéine de pleine longueur en présence d’un ncAA et d’une protéine tronquée en l’absence de ncAA indiquerait que le système ncAA-RS/ARNt(CUA) a été capable de reconnaître le codon TAG et d’incorporer le ncAA, mais pas les acides aminés endogènes.
La qualité d’une étude sur les protéines avec ncAA est définie par ce que l’on peut comprendre de la structure ou de la fonction de la protéine, ou par la nouvelle capacité conférée par la présence de ncAA dans la protéine. Étant donné que les ncAA élargissent le potentiel chimique limité des résidus d’acides aminés, une grande variété d’études et d’applications deviennent possibles pour explorer la nature à la fois sensible et résistante des protéines lorsqu’on utilise des ncAA. Selon l’emplacement et le type de substitution d’acide aminé, un large éventail d’effets sur la stabilité et/ou la fonction de la protéine peut se produire : les deux peuvent ne pas être touchées, la stabilité peut ne pas être touchée mais l’activité détruite, ou encore la protéine peut même ne pas se replier correctement, détruisant ainsi la fonction. Il a même été démontré que certains ncAA placés dans le site actif de l’enzyme améliorent la fonction de l’enzyme en modifiant l’électrostatique de la liaison ou de la catalyse. Si la stabilité et la fonction de la protéine ne semblent pas très perturbées par l’ajout du ncAA, les propriétés chimiques du ncAA peuvent être utilisées pour fournir des données pertinentes sur les états structurels de la protéine. Le tableau 1 présente des exemples de différents ncAA et d’études d’application.
Tableau 1. Quelques-unes des familles de ncAA et leurs applications.
Que la fonction de la protéine soit améliorée ou apparaisse plus clairement, ou que la structure et la dynamique de l’enzyme soient mieux comprises, l’utilisation des ncAA a permis de réaliser un plus grand nombre d’études sur les relations entre la structure et la fonction des protéines.
| Catégorie de ncAA | Exemple de structure | Applications |
| Photoréticulation | 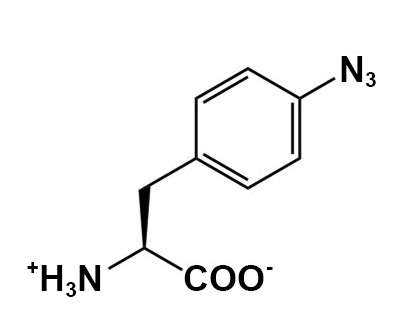 |
Aperçu des interactions entre protéines in vivo |
| Ligature bi-orthogonale |  |
Conjugaison des fluorophores; fonctionnalisation de surface |
| Sondes d’évaluation de la taille et de la polarité | 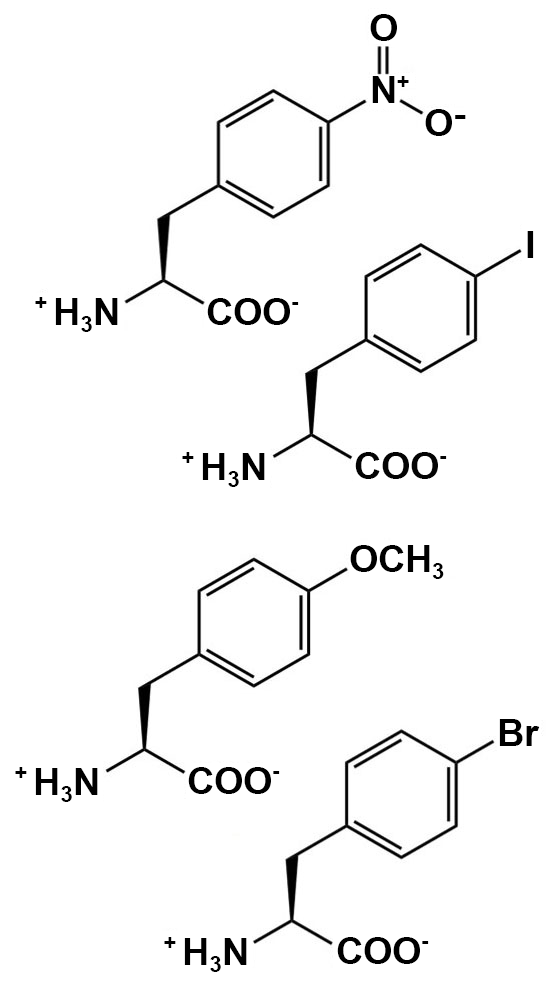 |
Altération du remplissage, de l’effet stérique et des autres interactions pour sonder les relations entre la structure et la fonction |
| Sonde du pH | 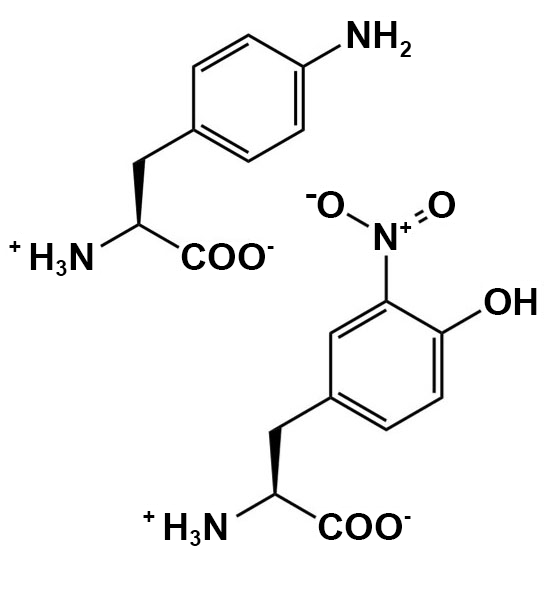 |
Ajout, suppression et modification des interactions de liaison hydrogène pour étudier les relations entre la structure et la fonction |
|
Réflexions approfondies sur les réflexions approfondies Tout au long de ce manuel, vous verrez des remarques intitulées « Réflexions approfondies » portant sur différents sujets. Elles ont été ajoutées pour vous aider à mieux comprendre votre projet et les techniques que vous utilisez, et pour vous fournir des éléments à prendre en compte lors de la conception de votre expérience, de l’analyse de vos résultats et de l’exécution des différentes étapes de ce laboratoire. Ces suggestions ne sont pas exhaustives; elles ont pour but de vous aider à réfléchir de manière critique à ce que vous faites et de guider votre conception et votre processus de réflexion. Nous nous attendons à ce que vous ayez lu, réfléchi et répondu à ces questions par vous-même avant de demander de l’aide à l’enseignant ou à l’assistant d’enseignement. |

